《细胞》杂志以封面故事形式重点关注了本项研究
人力背负起的癌基因星球,燃炸天
早在2005年,一群科学家提出,要通过基因组学分析技术把人类全部癌症的基因组描绘出来,从而更好地了解癌症。十余年的时间,顶尖科学家们完成了常见的33种人类癌症、11000余例病例基因数据的收集,癌症基因组图谱(TCGA)[1]的出现,为我们展开了癌症研究的新天地。
然而数据库的完成并不是一切的终结。250万GB的海量数据,其中就隐藏着人类杀手的真正命脉所在,如何抓住它们、并应用在临床研究和治疗上,这才是真正的挑战。
就在今天,一项新的研究成果给我们带来了癌症研究的全新视野!顶尖学者们对癌症基因组图谱的数据,以及癌症转录组学、蛋白质组学、甲基化组学、临床数据进行了综合分析,挖掘出其中的有效信息,成功绘制出泛癌症图谱(Pan-Cancer Altas)[2]!该图谱对癌症如何产生、在何处产生、为何产生进行了深入的剖析,毫不夸张地说,这就是人类癌症研究的“谷歌地图”!
泛癌症图谱数据报告分为三大部分、26篇科研论文,分别发表在《细胞》及其旗下《细胞报告》、《癌细胞》、《细胞系统》、《免疫》等顶尖医学杂志上。每部分由一篇旗舰论文叙述该部分主要研究成果,其他辅助论文则从各角度进行了深入的解释。
Cell-of-origin patterns 细胞起源模式
随着诊疗技术的不断发展,科学家逐渐发现,传统的组织病理学分析已经不足以描述癌症的类型,部分肿瘤内部仍旧存在较大的异质性。泛癌症图谱根据分子特征对人类癌症类型进行了重新分类,为科研和临床试验设计带来了新的见解。

本部分的旗舰论文来自美国北卡罗来纳州立大学教堂山分校Katherine A. Hoadley教授和Van Andel研究所Peter W.Laird教授团队。研究者们采用一种名为iCluster[3]的算法,对33种癌症、11286份肿瘤样本的染色体非整倍性、DNA甲基化、mRNA、miRNA和蛋白质数据进行了整合分析[4]。
根据分子特征,研究者们将解剖学上的33种癌症重新归类为28种亚型,针对某一种分子亚型,其中竟能包括传统解剖学意义上的25种癌症,真是太颠覆了!
在癌症的发展过程中,癌细胞的形成和分化程序、原始细胞的表观遗传状态、病原体和炎症等致突变因素,都会影响癌细胞的基因组,这就造成我们认为的同一种癌症,可能在分子水平上具有很大的差异。本项研究中,研究者发现,仅有1/3肿瘤表现出了同质性,其余2/3均有不同程度的异质性。
同种癌症有不同的分子特征,不同种癌症却具有同样的分子特征,这可能会导向新的治疗方案。Katherine A. Hoadley说道:“一直以来,肿瘤的位置是我们确定治疗方案的主要方法,而这项研究对我们了解不同肿瘤类型之间的关系是有帮助的。如果肿瘤具有基因多样性,那么我们应该重新思考对待它们的方式。”[5]
据研究者估计,以分子特征作为基准,至少将会有1/10癌症患者需要重新分类,可能需要新的治疗方案。
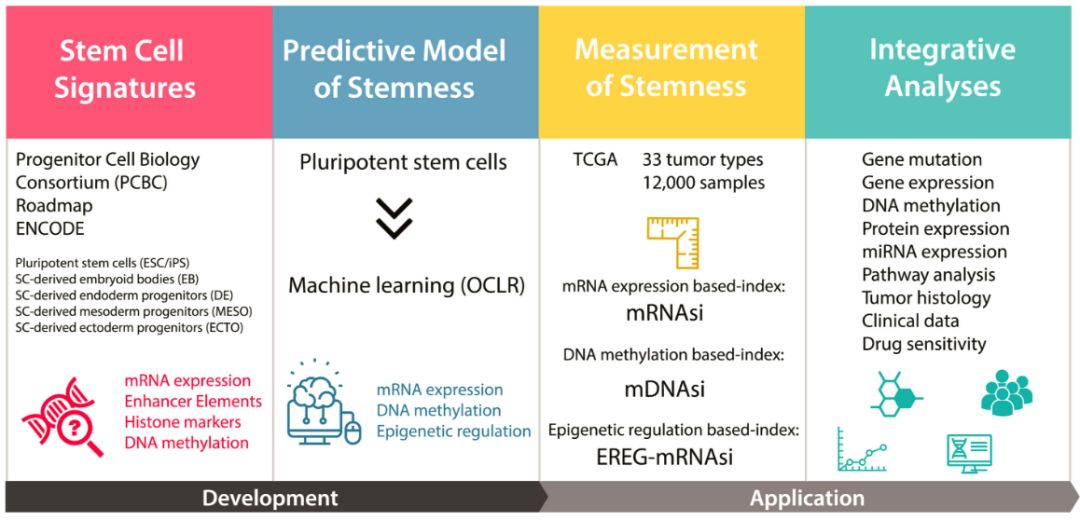
同样发表在《细胞》上的另一篇研究,则是利用机器学习算法测定癌细胞的干细胞样特征,以此鉴定可能的新靶点[6]。
在癌症的进展中,癌细胞会逐渐通过去分化获得祖细胞和干细胞样特征。研究者开发了一种对数回归(OCLR)机器学习算法,通过对细胞转录组和表观遗传学特征的分析评估癌细胞去分化程度。研究者意外发现,癌细胞的去分化程度与PD-L1水平以及免疫微环境有关,而且转移型癌细胞的去分化程度更高。
本部分的其他4篇论文则是对乳腺癌、胃肠腺癌、鳞状细胞癌和肾细胞癌的分子特征进行了深入的分析。
Oncogenic Processes 致癌过程
万物发展皆须一定的过程,癌症也是如此。从一个小小的基因突变到张牙舞爪的癌细胞大军,其中包含无数的的分子水平过程。泛癌症图谱对致癌突变与基因、蛋白各分子层面的作用进行了详尽的分析,让我们更清楚地“看见”癌症如何生长。这部分内容揭示了生殖细胞和体细胞突变在癌症进展中的作用,并论述了突变对细胞信号传导和免疫细胞组成的影响。
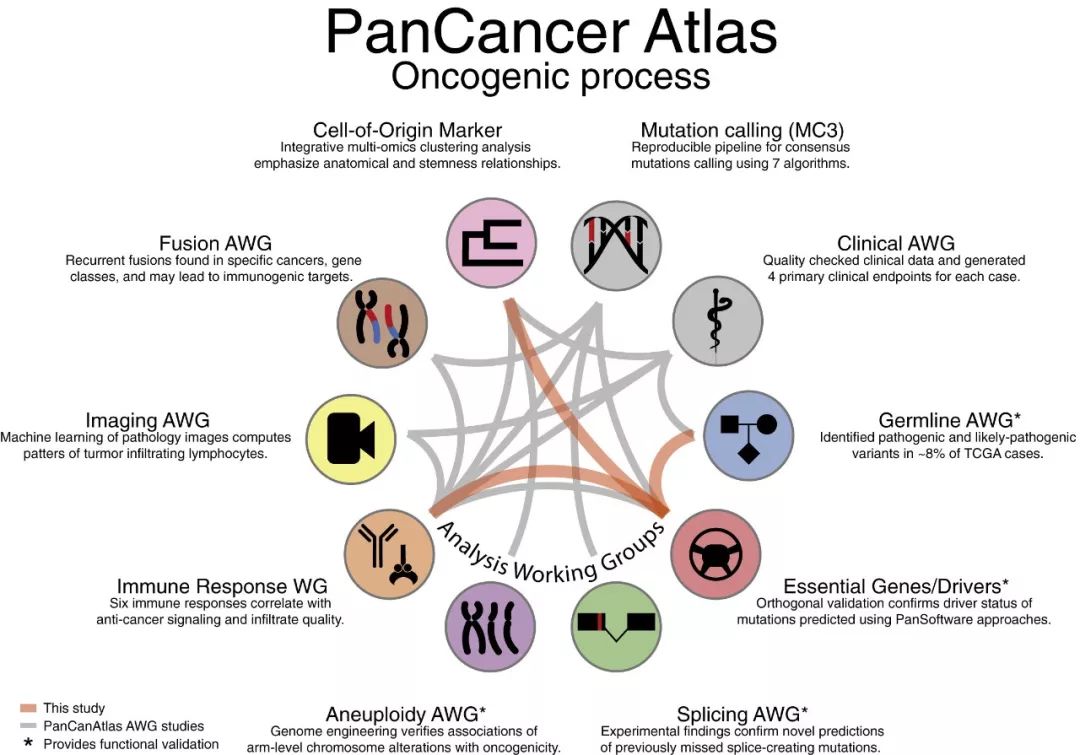
本部分的旗舰论文来自哈佛医学院、贝勒医学院与华盛顿大学的合作团队。研究者们从肿瘤发生过程中三个方面进行了分析,包括:(1)体细胞与生殖细胞突变在肿瘤发生中的相互作用;(2)肿瘤基因组和表观遗传组对转录组和蛋白质组的影响;(3)肿瘤与微环境之间的关系[7]。研究者将大量数据进行了整合,阐明了复杂的致癌过程。
在半个世纪的“癌症之战”中,DNA测序给预防和治疗带来了新的希望,靶向抑癌因子突变、融合蛋白、免疫应答等新型疗法逐渐走进视野。生物医学研究正逐渐离开对肿瘤的单一评估,多组学将成为科研的主流。
在本部分的其余12篇论文中,研究者们详细论述了癌症相关的数百种生殖细胞突变、融合基因、免疫组化模式、淋巴细胞浸润模式、增强子活性、染色体非整倍体、长链非编码RNA(IncRNA)、剪切突变等分子特征与癌症发展的关系。
这些复杂的相互作用过程表明,我们应当在更广阔的视野下评估癌症,这将导致临床和科研的巨大变化。
Signaling Pathways 信号通路
控制细胞生长、凋亡和细胞周期的信号通路改变是癌症的常见标志。泛癌症图谱对不同癌症中信号通路的改变程度和机制进行了分析,揭示了这些过程在一系列癌症中的相似性和差异性。研究涉及了经典癌症通路Myc、Notch、β连还蛋白/WNT通路等,这将有助于开发新的联合疗法和个性化治疗。

本部分的旗舰论文来自纪念斯隆凯特琳癌症研究中心、Dana-Faber研究所和洛桑大学。研究者对9125个肿瘤样本中的突变、拷贝数、mRNA、基因融合和表观遗传数据综合分析,探索了十种经典通路中基因突变的模式和机制,包括细胞周期、Hippo、Myc、Notch、Nrf2、PI3/Akt、RAS、TGFβ、p53和WNT通路[8]。
根据通路变化的情况,研究者把33种癌症分为64种亚型,确定了各通路之间协作和排斥的模式。研究者发现,89%的肿瘤至少存在一种通路改变,而且根据现有的靶向药物治疗方案,至少57%的肿瘤能够得到一种有效的治疗,30%的肿瘤响应多靶向治疗,很可能会带来新的联合治疗方案。
本部分的其他6篇论文则分别针对Myc、RAS、DNA损伤修复(DDR)、代谢变化、泛素、剪切因子等通路进行了泛癌分析。
我们犹豫了很久要怎样向大家说明这种数据库的存在意义,毕竟浩如烟海的数据,看起来远不如一种有效的新药来得实在。但是从另外一个角度来看,它丰富的内容给了科学家们无穷的研究素材。
癌症基因图谱的大量数据,使得研究者得以深究癌症中发生的分子变化,对癌症进行更有现实意义的分类,而且在其他原本没有预料到的方面也带来了无形的进步。
癌症基因组图谱是一项伟大的工程,它的完成离不开贯穿全美和加拿大的20余所科研院校顶尖科学家的通力合作。这使得科研成果达到了个人实验室研究所无法达到的高度,并引导了此后一批大型科研项目的合作模式。
在研究过程中,对大批量基因数据的处理将计算机方法引入生物科研领域,间接促进了计算生物学的发展,也为其他人类疾病的基因组学研究打下了基础。
此外,癌症基因组图谱数据库面对科学界和一般群众开放,任何人只要想要,都能够接触到这些数据。这一方面令原始数据能够得到多角度、多层次的分析,产生新的科研成果;另一方面,科研成果的共享也促进了数据文件的标准化,对科学界整体的发展来说,无疑是极为有利的。
而这一切,都是建立在科学家们以人力绘制出的每一个细节之上。
十余年前,癌症基因组图谱计划开始的时候,以当时的科技水平,这看起来甚至是一项不可能完成的任务。
如今,我们已经将癌症的“谷歌地图”握在手中。
泛癌症图谱并不是数据的终点,相反,它将会成为另一块基石,帮助研究者在特定癌症领域、以及更广阔的生物医学领域走得更远。

http://www.cell.com/pb-assets/consortium/PanCancerAtlas/PanCani3/index.html
官方网站查看更多研究论文信息
泛癌症图谱的网站首页,以大号字体写着“Welcome to the Pan-Cancer Atlas”,这看起来好像是在说
欢迎来到新世界
编辑神叨叨
清明节的办公室静悄悄~这有一只糕在写稿~虽然研究很重要~但是26篇论文也太多liao~
参考资料:
[1]http://www.cell.com/cell/fulltext/S0092-8674(18)30391-X
[2]http://www.cell.com/pb-assets/consortium/PanCancerAtlas/PanCani3/index.html
[3] Shen, Ronglai, Adam B. Olshen, and Marc Ladanyi. "Integrative clustering of multiple genomic data types using a joint latent variable model with application to breast and lung cancer subtype analysis." Bioinformatics 25.22(2009):2906-2912.
[4]http://www.cell.com/cell/fulltext/S0092-8674(18)30302-7
[5]https://www.genomeweb.com/sequencing/tcgas-pan-cancer-atlas-papers-examine-clustering-tumors-key-alterations#.WscdXt-Wa02
[6]http://www.cell.com/cell/fulltext/S0092-8674(18)30358-1
[7]http://www.cell.com/cell/fulltext/S0092-8674(18)30313-1
[8]http://www.cell.com/cell/fulltext/S0092-8674(18)30359-3
[9]http://www.cell.com/cell/fulltext/S0092-8674(18)30380-5
[10]https://www.nih.gov/news-events/news-releases/nih-completes-depth-genomic-analysis-33-cancer-types
[11]http://www.cell.com/cell/fulltext/S0092-8674(18)30381-7

















