2月2号,science上发表了题为《Functional traits—not nativeness—shape the effects of large mammalian herbivores on plant communities》的论文(Lundgren et al. 2024),论述了大型植食性动物对植物群落的影响主要取决于其功能属性。

抛开科学问题不说,这是一篇典型的Meta分析论文,通篇采用Meta分析的套路完成,值得学习。该研究也是针对所有其检索到的进行了大型植食性动物隔离处理的案例研究进行了数据提取,通过构建效应值和随机(混合)效应模型对动物隔离措施对植物群落的影响进行非常系统的研究,同时分析了这种影响随动物的各种功能属性的变化关系,研究非常全面。
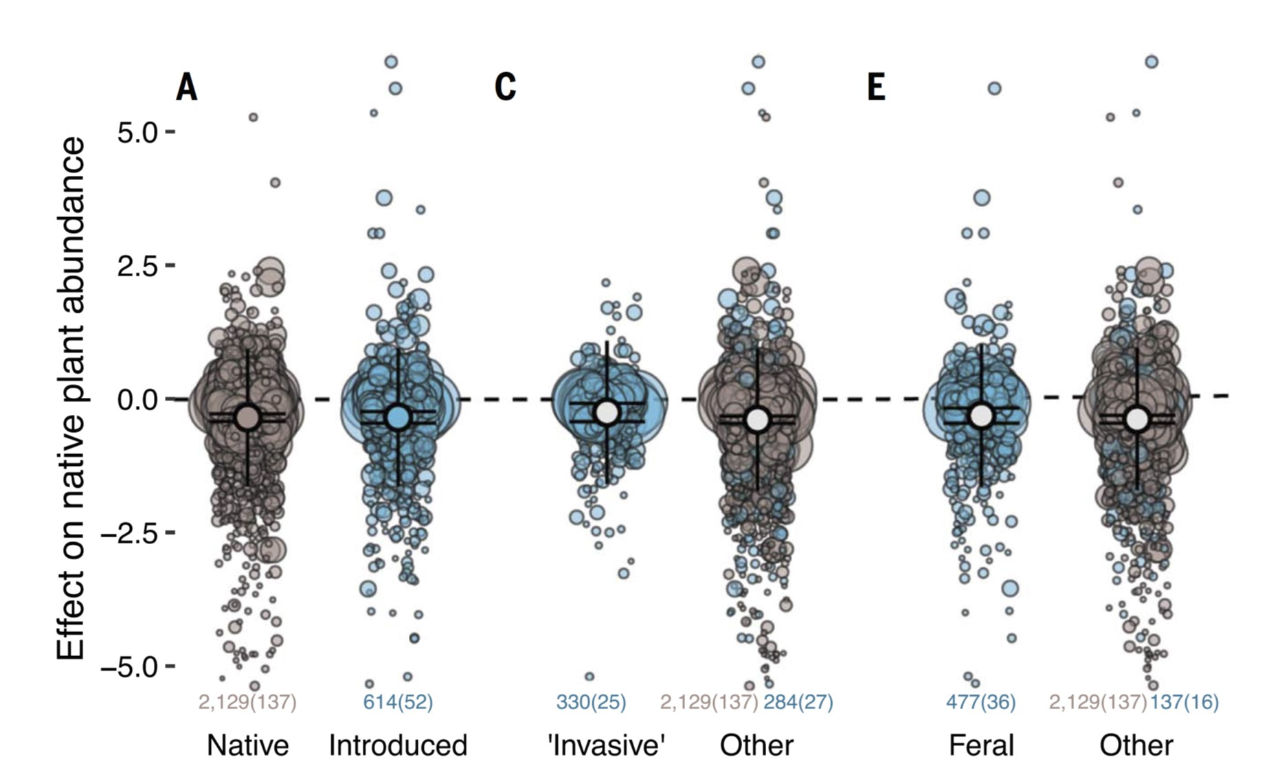
从Meta分析的技术层面上,有几个要点:
1)采用Hedges’ g作为效应值,同时计算了对应的案例内方差。
2)采用metafor下面的rma.mv和随机效应模型计算累计效应,采用混合效应模型进行影响因素分析,且所有的模型都去掉了截距项。
3)多重比较采用compound包下的glht函数完成
4)重点:由于动物隔离实验的结果具有时间序列上的重复测量特征,所以数据分析中把时间自相关结构(ar1型)加入到了模型之中(rma.mv, struct函数)。并对比了是否考虑这一结构,模型拟合度是否有改善。
5)模型中考虑多个随机效应,如地点,文献,物种
6)参数估计采用最大似然法(因为要进行模型对比)
7)采用漏斗图进行了发表偏爱性诊断
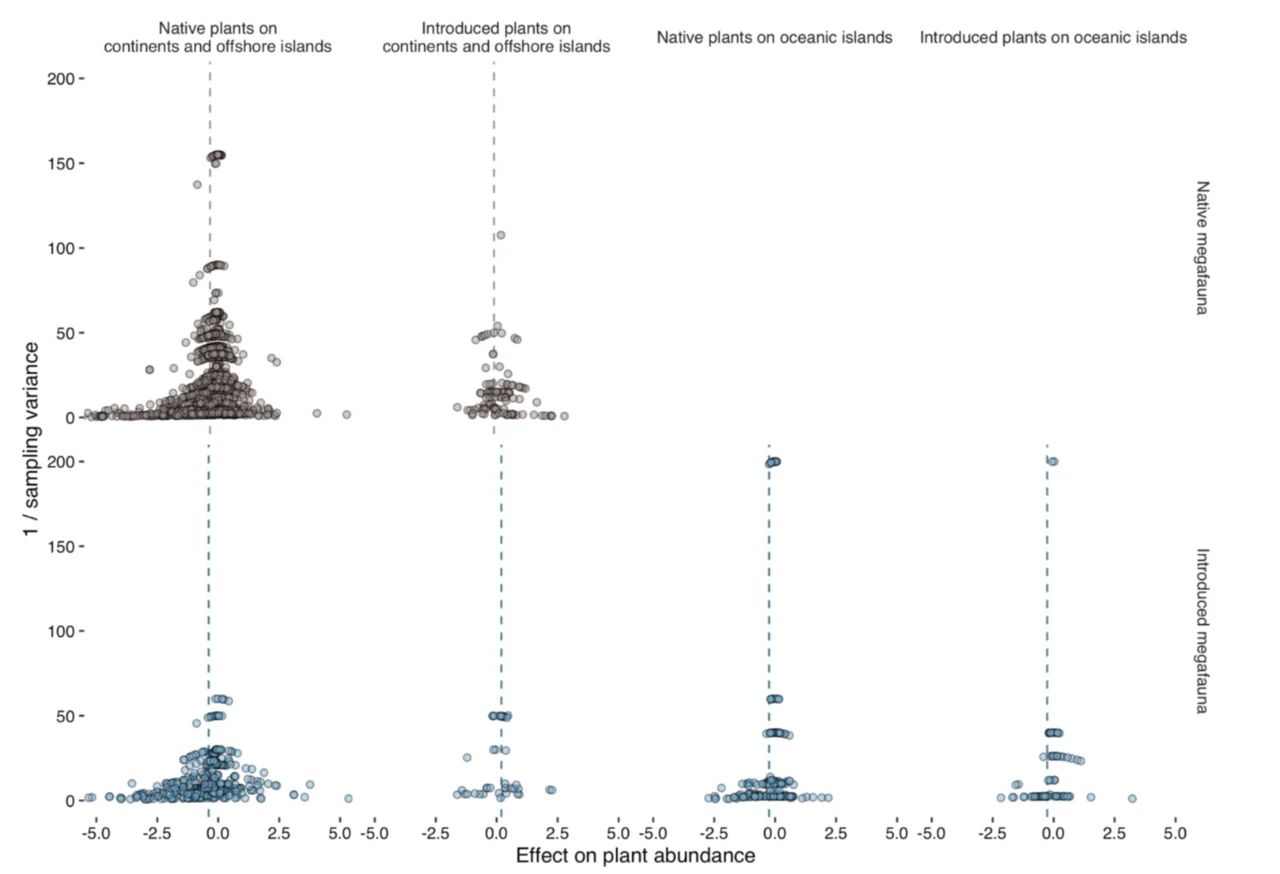
发表偏爱性的漏斗图检验
以其中一个模型为例,其模型代码为:
m <-
rma.mv(yi_smd ~ Herbivore_nativeness + MAP - 1,
V = vi_smd,
random = list(~1 |
Citation/species_response_tag,
~1 |
site_id,
~time_series_clean
| experiment_id),
struct =
"AR",
method =
"ML",
test =
"t",
dfs =
"contain",
data = dat)
其中~time_series_clean | experiment_id即为设置重复测量的的设置,struct = "AR",为时间自相关类型的设置。其所有模型(一共包括449个模型)都是采用类似设置完成。该研究附件里也提供了数据和代码,大家可以自行下载学习,这篇文章也是大家学习meta分析的理想案例,这里也向作者公开其数据和代码表示衷心感谢!
参考文献:
Lundgren, E. J., J. Bergman, J. Trepel, E. le Roux, S. Monsarrat, J. A. Kristensen, R. Ø. Pedersen, P. Pereyra, M. Tietje, and J.-C. Svenning. 2024. Functional traits—not nativeness—shape the effects of large mammalian herbivores on plant communities. Nature 383:531-537.

















