1.蛋白互作网络
STRING数据库,全称为Search Tool for Recurring Instances of Neighbouring Genes,可用于蛋白相互作用分析(Protein-protein interaction, PPI),使用String可构建互作网络。
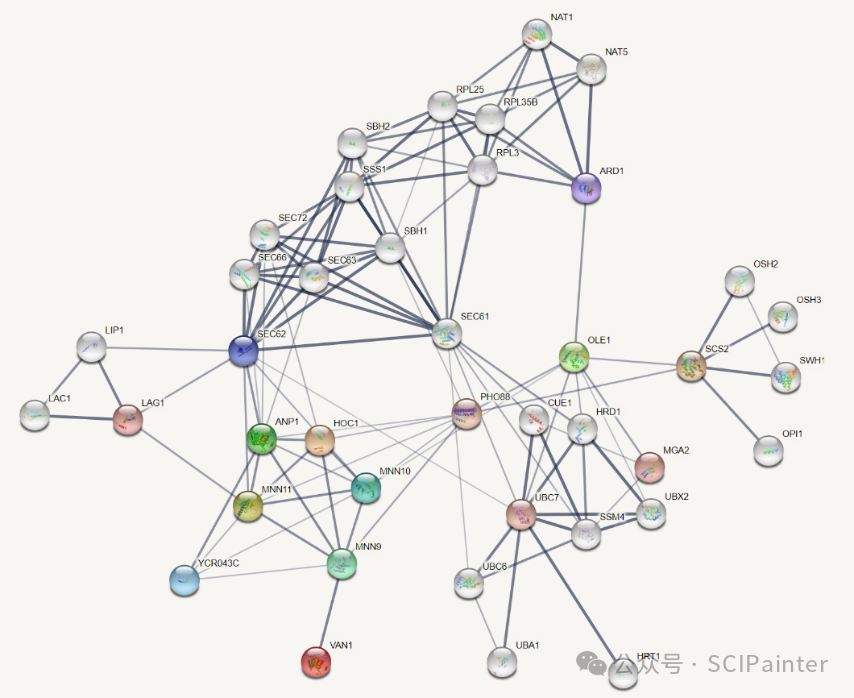
详细教程链接:
《如何进行基因、蛋白互作网络分析?》
2.共发生网络图
除了社交网络分析,在自然科学领域,特别是微生物生态学相关的科研人员,通常使用igraph包和Gephi做微生物群落的共发生网络分析(Co-occurrence network analysis)。

详细教程链接:
《如何绘制漂亮的网络图?》
3.环状排序网络图
使用STRING数据库完成蛋白互作网络的构建后,用Cytoscape软件打开,只需通过Layout菜单下Attribute Circle Layout的Degree属性进行布局,如下,即可得到排序后的环状网络。
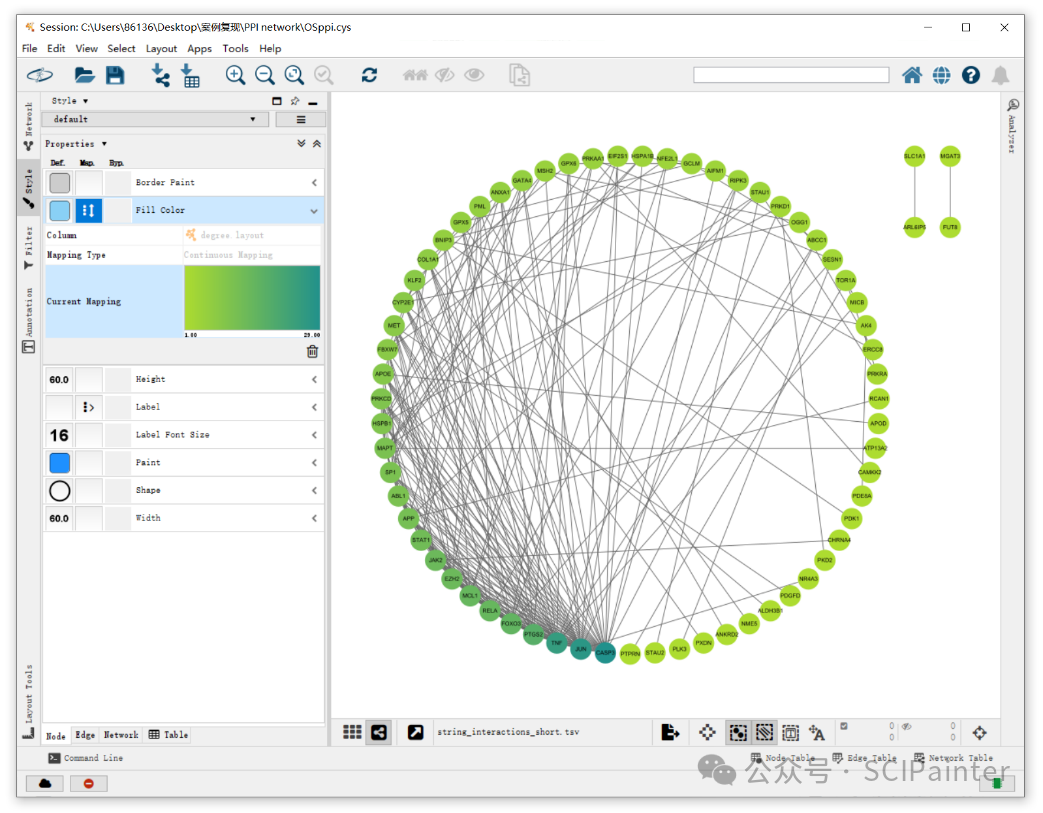
详细教程链接:
《如何构建排序的环状蛋白互作网络图?》
4.转录因子互作网络
得到Hub基因后,接下来可以使用Network Analyst进行转录因子的预测和互作网络的构建。
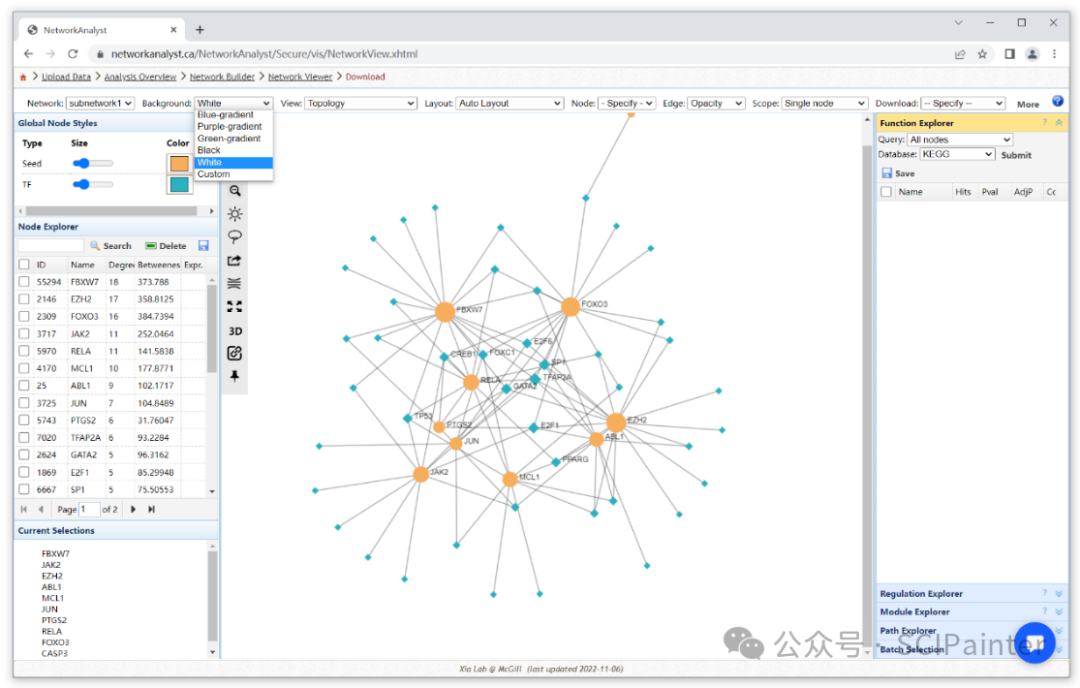
工具的链接:
https://www.networkanalyst.ca/
详细教程链接:
《如何预测转录因子并构建互作网络?》
5.细胞通讯网络图
细胞通讯网络图数据的整理很简单,source、target列分别对应不同的细胞亚群,count列对应亚群之间显著富集的配受体对数目,然后另存为表格文件,然后导入Cytoscape进行可视化即可。
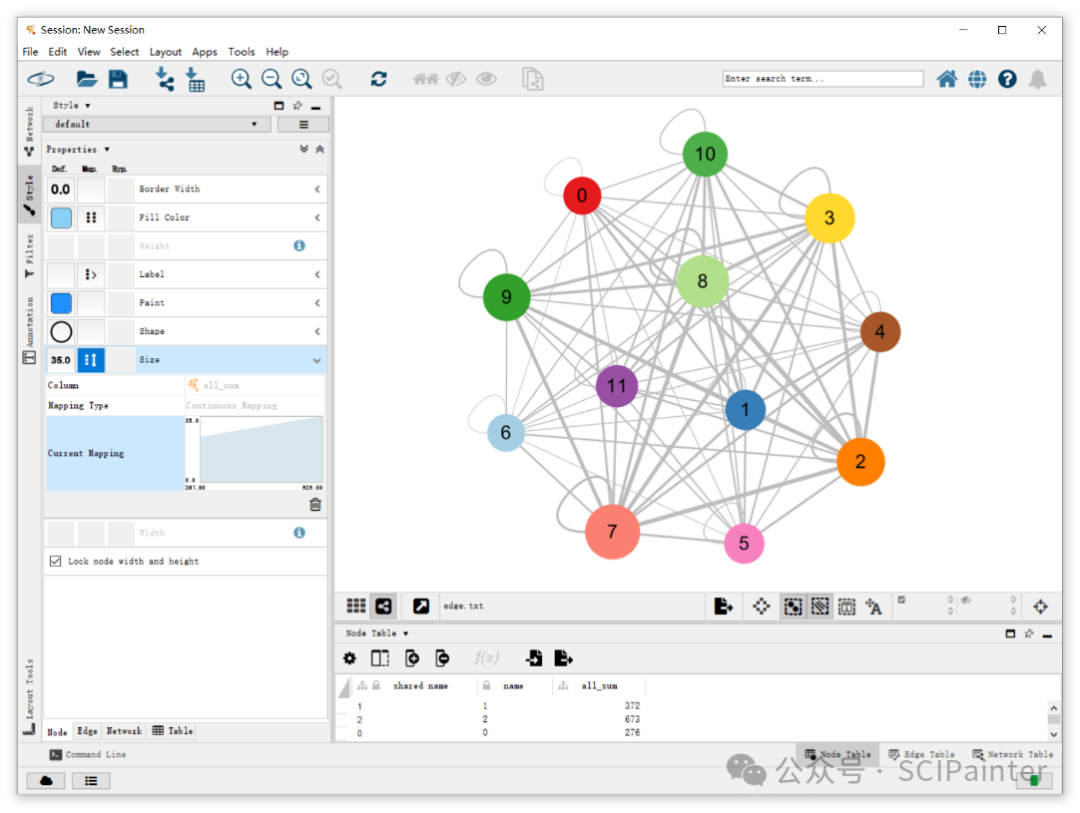
详细教程链接:
《如何绘制好看的细胞通讯网络图?》
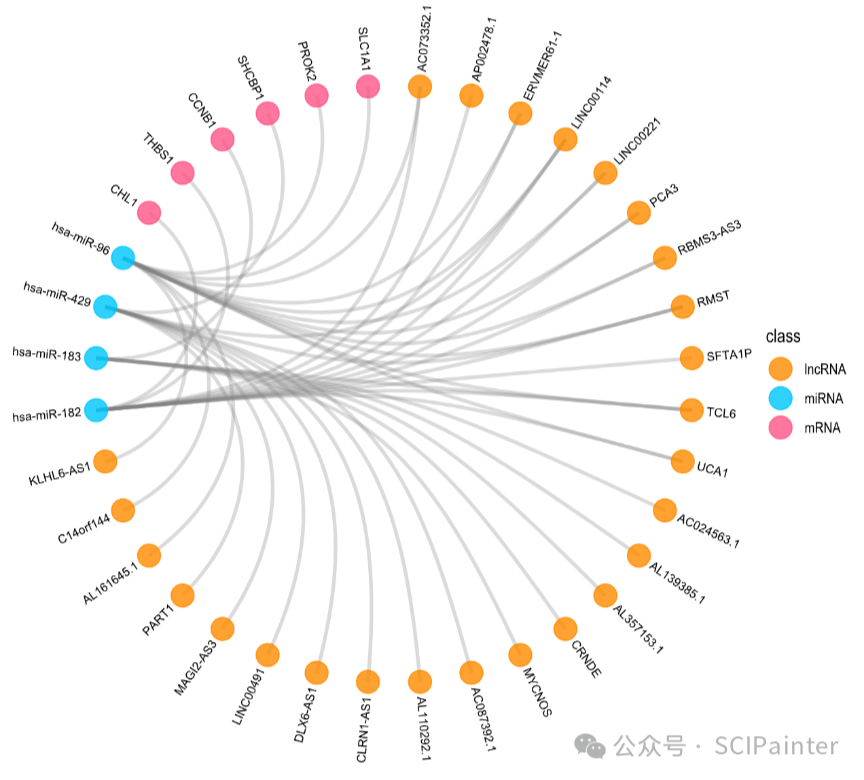
6.环状ceRNA网络图
环状网络绘制方法其实比较简单,除了使用Cytoscape软件,使用ggraph包可实现网络图的绘制。
详细教程链接:
《网络图画成这样,是什么水平?》
7.网络韦恩图
网络韦恩图,用以展示不同分组之间共有或特有的基因,特别是当分组元素数量较少时,数据展示效果更加直观。
详细教程链接:
《如何绘制各种高颜值的网络韦恩图?》
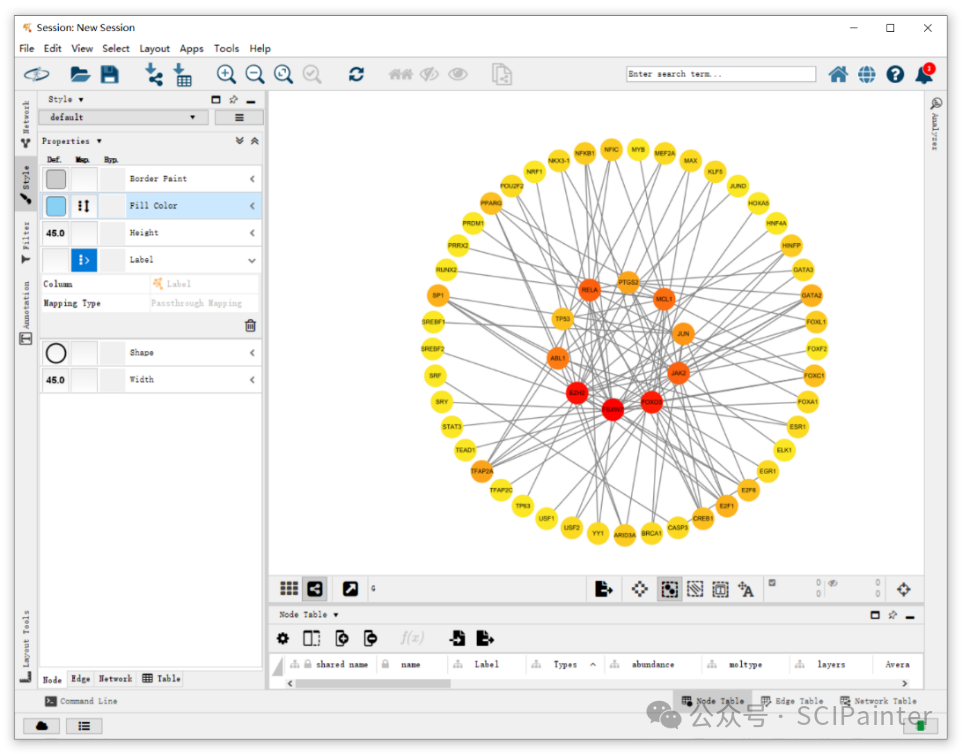
8. 圈圈网络图
通过Cytoscape软件的Layout Tools,可修改选中节点的布局(Layout),比如对网络图进行缩放(节点大小不变)、旋转以及对选中的节点进行对齐、均匀分布、堆叠等。按住Ctrl键框选节点,将其调整成环形,如下。
详细教程链接:
《如何绘制蛋白互作“圈圈”网络图?》
9.相关性网络热图
R包aPEAR可直接解析clusterProfiler包的富集分析结果,分析通路的基因列表、检测冗余重叠基因集群并绘制网络图,效果如下。
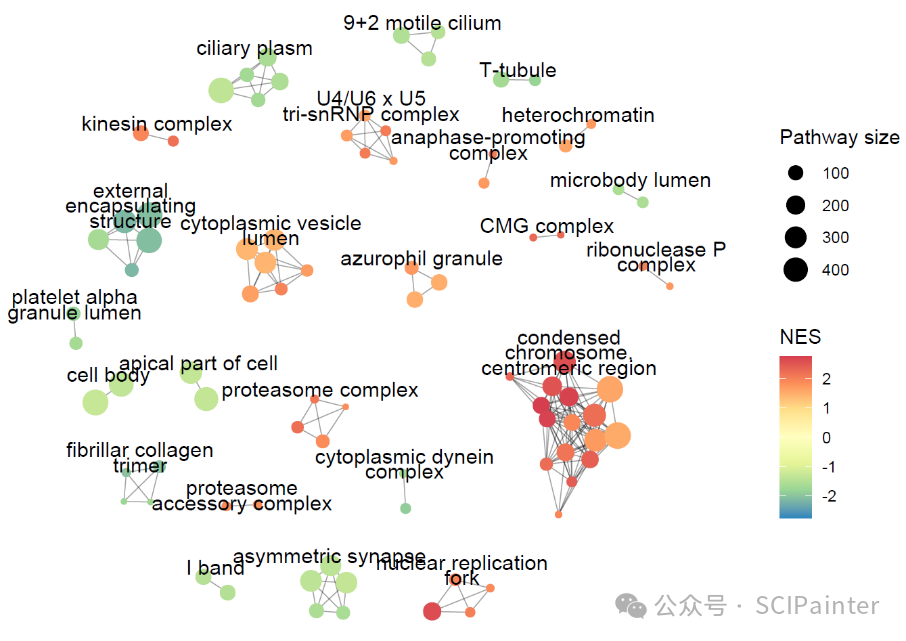
详细教程链接:
《如何绘制功能富集网络图?》
10.相关性网络热图
网络热图,应用于多组学关联分析,可展示组学内(热图)和组学间(网络图)的相关性结果。
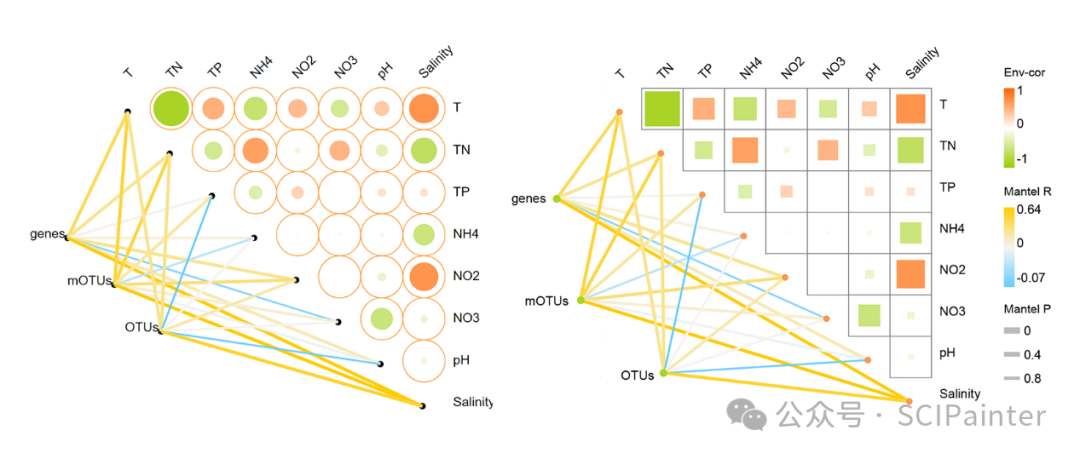
工具链接:
https://www.omicshare.com/tools
详细教程链接:
《推荐一个绘制Science网络热图的在线工具》

















