2024年诺贝尔生理学或医学奖授予两位发现microRNA的科学家Victor Ambros和Gary Ruvkun。这次诺奖使学界的视线再次聚焦到microRNA。microRNA作为转录后调控的重要一环,研究其生理学功能、分子机制、对人类疾病研究和治疗有重要意义。
本文聚焦免疫系统中microRNA的发现历史和最新进展,并尝试探讨该领域尚未解决的问题和发展方向。

1
microRNA的发现
20世纪70年代,麻省理工大学的Bob Horvitz实验室鉴定到了大量线虫的遗传突变。他的博士后Victor Ambros针对线虫的基因做了大量遗传学分析,发现两个有重要功能的基因,Lin-4和Lin-14。
1982年,另一位博士后Gary Ruvkun加入了Bob Horvitz实验室,参与了Lin-4和Lin-14的探索。1993年,两人在Cell上发表了背靠背的文章,揭示了Lin-4编码了一种微小RNA,可以转录后调控Lin-14编码的蛋白表达水平[1-2]。这是第一次揭示这种非编码微小RNA介导的全新的基因调控范式。
但是,有一个问题一直困扰着这两位科学家,Lin-4介导的调控是否是线虫独有的,而非大量存在于其它物种尤其是高等动物体内的重要调控方式? 1993-2000年间,Ambros实验室做了大量工作去寻找类似于Lin-4的微小RNA,但是徒劳无功。他自己也因此而离开了哈佛大学,加入达特茅斯学院。他的实验室也迫于经费短缺而在eBay上购买二手仪器设备。直到2000年,Ruvkun实验室成功鉴定到了另一个microRNA发现史上极为重要的分子,Let-7。尽管一开始Let-7也是在线虫中发现的,但是他们很快发现了Let-7进化上的保守性,并在包括人类和果蝇在内的许多物种中都鉴定到了Let-7[3-4]。
从此,microRNA的发现和研究开始进入黄金时期,成百上千种microRNA在不同物种中被发现。越来越多的研究报道其在生理学上,尤其是高等动物的发育和分化中,发挥着非常重要的作用。
Ambros在2008年回顾这段历史时,描述了在得知Let-7的发现及其在进化中的保守性之后的激动心情和这一发现的划时代意义:“在2000年的秋天我读到Gary 他们关于Let-7的文章之后,我凝视窗外十多分钟,重新梳理我对这个宇宙的认知。现在,我们终于明白了Lin-4和Let-7是一个进化中古老的调控分子家族的成员,因此动物中肯定还存在其它的类似RNA亟待发现[5]。
(“After reading their paper in the autumn of 2000, I had to set aside 10 minutes to stare out the window and reorganize my view of the universe. Now we knew that lin-4 and let-7 RNAs were members of an evolutionarily ancient class of regulatory molecules, and so it was finally obvious that there must be other RNAs like them yet to be discovered in animals”)
2
microRNA与免疫系统
2.1 大发现的前夜
2000年Let-7的发现点燃了科学家对发现和研究microRNA的兴趣。但是microRNA在哺乳动物中发挥何等作用,在复杂系统中如何调控基因表达,依然需要遗传学证据[6]。
免疫系统,尤其是后天免疫系统,是高等动物进化出的一套复杂且精细的防御系统。2003年左右,免疫学家们开始注意到microRNA在免疫系统中可能具备重要的调控作用,一些实验室开始构建Dicer 以及单个microRNA基因敲除小鼠。与此同时,在各种人类和小鼠组织中对microRNA进行检测显示其表达具有明显细胞和发育阶段特异性[7],一些microRNA在肿瘤组织中也有高表达[8-10]。这些证据提示microRNA可能发挥重要的调控作用。一些实验室采用骨髓重构或者过继回输的方式构建microRNA过表达小鼠,在免疫系统中观察到明显表型[11]。但是,这些发现亟待通过构建分析microRNA基因敲除和过表达小鼠来证实其在免疫系统中的功能以及研究它们发挥作用的分子机制。
2.2 早期研究
Dicer是产生成熟microRNA的关键加工蛋白[12-13]。Dicer缺失会造成绝大多数microRNA成熟出现问题,从而表达缺失。早期研究发现条件性敲除Dicer会导致造血系统发育以及B 和T淋巴细胞早期发育出现明显缺陷[14-16]。科学家们推测单个microRNA基因也可能在免疫系统中发挥重要作用。基于这一推测,一些实验室开始构建特定microRNA敲除小鼠。同时,针对肿瘤中高表达的microRNA构建转基因小鼠以研究这些microRNA过表达是否具有促癌作用。是否所有当时鉴定到的microRNA都有强大功能呢?
Klaus Rajewsky、Tyler Jacks、Martin Turner、Carlo Croce等实验室根据进化中的序列保守性、组织特异性、以及肿瘤中高表达等条件,将目光投向后来为我们熟知的几条在免疫学上具有重要功能的microRNA,包括miR-17-92、miR-155以及miR-150。
2.3 miR-17~92
miR-17~92最早于2004年在B细胞淋巴瘤病人样品中鉴定出来[17],是一个较大的microRNA基因家族,包括miR-17~92、miR-106a~363和miR-106b~25三个在基因组不同位置的同源基因簇,编码4个miRNA亚家族(miR-17、miR-18、miR-19和miR-92亚家族),共计13种microRNA。实验证明在Myc转基因小鼠中通过病毒感染过表达miR-17~92可以加快淋巴瘤形成[18]。Rajewsky实验室博士后肖昌春利用当时已经成熟cre-loxp系统构建了在淋巴细胞中特异表达的miR-17~92转基因小鼠,并发现这些转基因小鼠会自发产生脾脏和淋巴结肿大,淋巴细胞活化增殖,免疫系统紊乱,从而导致转基因小鼠提前死亡。这些结果提示了miR-17~92在淋巴细胞里面发挥了重要的作用[19]。
同一时期,Jacks实验室博士后Andrea Ventura构建分析了miR-17~92家族的三个基因簇敲除小鼠。他发现在全身敲除的情况下,小鼠胚胎发育有明显缺陷,敲除小鼠B细胞无法正常发育。同时,他的研究表明miR-17~92家族敲除小鼠表型主要来自于miR-17~92基因簇敲除,其他两个基因簇的功能较弱[20]。这两个实验室通过研究miR-17~92敲除和过表达淋巴细胞发现Bim和Pten是miR-17~92调控的主要靶基因。在miR-17~92敲除细胞中删除Pten和Bim的一个基因拷贝可以很大程度上回补miR-17~92敲除导致的表型。
2.4 miR-155
miR-155是一种在淋巴瘤中有高表达的microRNA并且在特定免疫细胞群中高表达[9]。2007年,Rajewsky实验室和Bradley实验室构建分析了miR-155敲除和转基因小鼠,在Science上背靠背发表了两篇文章揭示miR -155在免疫系统中发挥重要作用,尤其是在生发中心反应中有着重要功能[21-22]。敲除miR-155导致生发中心B细胞减少,过表达则导致生发中心B细胞增加。敲除miR-155也会造成T细胞分化和细胞因子分泌缺陷。之后不久,Turner实验室的研究进一步揭示了在B细胞中miR-155主要通过调控Pu.1控制抗体免疫应答过程[23]。而Nussenzweig实验室则采用构建过表达含有miR-155结合位点突变的Aicda转基因小鼠证明了Aicda也是miR-155调控B细胞功能的一个重要靶基因[24]。
2.5 miR-150
通过在小鼠免疫系统中检测microRNA表达谱[25],发现miR-150主要在成熟的静息态B和T淋巴细胞中表达,但是不在其前体细胞中表达。这种阶段特异性的表达模式可能意味着miR-150在B和T淋巴细胞中发挥特殊的功能。通过构建miR-150全身敲除小鼠和转基因小鼠,Rajewsky实验室博士后肖昌春发现miR-150的缺失会导致B1细胞增多,抗体免疫应答增强,而miR-150转基因小鼠则会导致淋巴细胞发育受阻[26]。分子机理研究发现miR-150主要通过抑制转录因子c-Myb的蛋白表达来实现其功能的,并且在遗传学水平上证明c-Myb杂合小鼠中c-Myb蛋白水平略微降低可以造成跟miR-150转基因小鼠类似的表型。
除了上述microRNA,在后续的研究中,免疫学家通过构建敲除小鼠研究确定了miR-142和miR-146a在适应性免疫调节上重要功能[27-28]。通过对以上microRNA敲除或者转基因小鼠的研究,这些实验室确定了microRNA介导的转录后调控在高等动物的生物学过程尤其是免疫系统中发挥了重要的作用。
同时,通过对microRNA结合靶基因的预测和构建杂合或者纯合靶基因敲除小鼠,为microRNA如何调控靶基因以及如何调控生物学过程提供了关键的遗传学证据,对理解microRNA在不同细胞类型中如何精准调控靶基因表达的分子机制具有重大意义。免疫学家还通过分析microRNA和mRNA表达谱以及CHIP-seq,探究了microRNA自身表达特点以及如何受到调控。2010年的一篇研究阐明淋巴细胞特异的microRNA是在转录以及转录后水平上受到严密的调控,也侧面说明microRNA是生物体内基因表达调控的重要一环[29]。
但是,受限于当时的技术手段,microRNA结合靶基因缺乏生化和分子水平上的实验验证。而且microRNA对大量潜在靶基因在mRNA和蛋白水平上的调控也缺乏定量分析。也应当注意到,在早期研究中,一些表达特异、高丰度、被预测为有功能的microRNA在后续的小鼠遗传学研究中表明并没有明显的免疫学功能,或者过表达有明显表型,但是敲除没有造成明显功能异常。比如miR-181[30],miR-210[31]以及miR-148[32-33]。肖昌春实验室构建了miR-148a以及同源microRNA(miR-148b和miR-152)敲除和转基因小鼠。对这些小鼠进行分析表明尽管过表达破坏了B细胞中枢耐受[33],敲除miR-148相关microRNA在免疫系统中没有发现明显表型(实验结果尚未发表)。因此,小鼠遗传学证据依然是microRNA功能研究的金标准,对microRNA研究有重要意义。
3
microRNA调控机制之争
随着越来越多功能重要microRNA的发现和研究,阐明microRNA调控靶基因表达的分子机制成为microRNA研究领域的焦点和热点。
2009年,David Bartel发表综述文章总结了microRNA 识别其靶基因的分子机制,考虑了microRNA seed region的匹配,UTR区域的上下游序列影响,microRNA以及其靶向mRNA的细胞内浓度等因素并阐明了这些因素如何影响microRNA对靶基因的抑制程度[34]。文章认为microRNA可以通过同时对很多mRNA的表达进行微调,从而实现对生理学过程的调控。也就是说,数百个靶基因表达水平的微小变化可以实现microRNA的强大功能。
同年,肖昌春和Rajewsky也发表一篇关于microRNA在免疫系统中调控机制的综述[35],对于microRNA如何通过调控其靶基因来调控生理学过程提出了“关键靶基因”这一理论。基于小鼠遗传学证据,他们提出了:一,microRNA对靶基因的调控有细胞类型特异性,特定细胞内的诸多因素会影响microRNA对靶基因mRNA的识别和调控。二,不同靶基因具有不同的剂量敏感性。microRNA是通过对为数不多的关键基因进行表达剂量的调控来实现其生理学功能。随后的一些研究,尤其是随着组学技术进步和Ago2 CLIP技术成熟,以及越来越多的遗传学证据表明,microRNA的生理学功能是通过对关键靶基因的调控来实现的。科学家们发现,在不同免疫细胞类型中,microRNA主要通过对为数不多的几个靶基因进行表达水平上的调控来实现其功能的。比如miR-155,在B细胞中主要是通过调控Pu.1, Aicda以及Smad5来发挥功能,在T细胞中则主要通过调控Socs-1, Sp1r1以及c-maf来发挥功能[36]。2018年,Rudensky实验室利用组学技术研究了miR-155在不同免疫细胞中结合的靶基因mRNA以及对靶基因mRNA表达水平的调控,从而在分子水平上证明细胞类型特异性对microRNA结合靶基因mRNA和调控它们表达的影响[37]。
另一个重要的microRNA是miR-17~92。Rajewsky和肖昌春实验室对其的系统性研究也提供了很多重要的见解。在分子机制上,尽管很多microRNA主要调节靶基因mRNA的降解,miR-17~92则主要调控靶基因的翻译。所以对mRNA和蛋白表达水平进行准确定量对理解microRNA调控机理非常重要。肖昌春实验室在免疫细胞各个发育分化阶段敲除和过表达miR-17~92[19,38-42],发现miR-17~92在不同免疫细胞以及它们的不同发育阶段具有不同的功能和关键靶基因。并且,miR-17~92在B细胞中的敲除和过表达可以实现对不同靶基因的调控。在功能水平上,在B细胞中敲除miR-17~92会导致抗体应答缺陷,过表达则导致形成淋巴瘤 [43]。2019年,Rajewsky实验室报道了miR-17~92关键靶基因Bim的主要miR-17~92结合位点突变小鼠[44]。该研究从靶基因角度探究了microRNA调控的细胞类型特异性和关键靶基因对microRNA功能的贡献。Bim基因上miR-17~92结合位点突变小鼠揭示了Bim的表达剂量对其介导miR-17~92功能的重要性,可以解释大部分miR-17~92敲除小鼠的表型,但也发现在B细胞发育阶段miR-17~92对Bim表达水平的调控并不明显,对B细胞发育也影响不大,而原因很可能是这个阶段Bim表达水平太高,超过了miR-17~92可以调节的阈值[45]。
4
总结
microRNA作为生物机体基因表达调控的重要一环,是目前发现的主要非编码RNA调控元件,对我们理解中心法则中RNA分子作为主要遗传信息分子的功能有着非常重要的价值。
随着对microRNA生理学功能和分子机制研究的深入,和越来越多小鼠和人类遗传学证据,研究和理解mRNA层面上复杂而精妙的调控已经成为解码生命科学的下一关键研究方向。而在抗击新冠疫情中初露峥嵘的mRNA疫苗也展现了RNA研究在转化应用方面巨大的潜力。mRNA已经成为一个关键的基因表达调控平台。mRNA的命运非常复杂,包括但不局限于修饰、编辑、翻译和降解,这才得以塑造出生命机体的复杂性和多样性。
以microRNA为例,下图展示了microRNA结合调控靶基因mRNA的命运受到多种顺式和反式作用元件的影响,包括mRNA自身的5‘和3’非翻译序列以及细胞类型特异的一些调控[46]。microRNA调控方式和作用也非常复杂,可能通过促进靶基因mRNA降解或者抑制其翻译来实现调控。甚至有研究认为microRNA可以促进mRNA表达水平上调[47]。因此,mRNA层面的调控机制还需要更深入更系统性的研究。
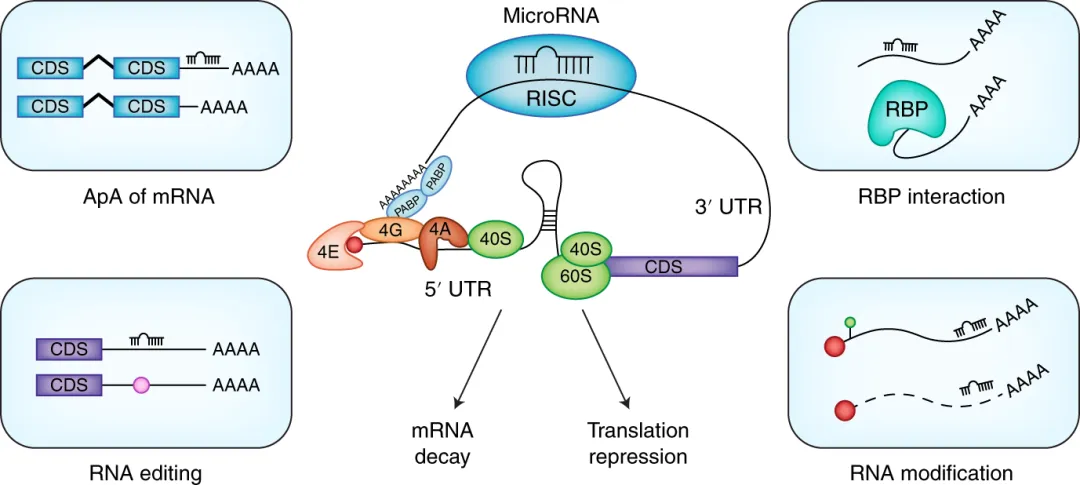
图片来源:Chen P, Liao K, Xiao C. Nat Immunol 19,
1040- 1042(2018).[46]
结合当今技术的发展,我们提出了系统性研究microRNA调控的范式。
一,利用RNA-seq、Ribo-Seq、和定量质谱全面分析microRNA对mRNA表达水平、翻译速率、和蛋白水平的影响。
二,在野生型和特定microRNA敲除小鼠的原代细胞中利用Ago-CLIP方法分析该microRNA结合的靶基因[48]。
三,利用CRISPR高通量筛选对鉴定到的靶基因进行功能分析,找到对microRNA功能有贡献的靶基因。
四,利用其他生化和组学实验方法分析顺式和反式作用原件,如polyA尾测序,CLIP结合位点上有共定位且和RISC复合物有互作的RNA结合蛋白, RNA的二级或者三级结构分析,以系统生物学思维构建更精准的microRNA调控靶基因的模型。另外,构建microRNA结合位点突变小鼠,尤其是那些在免疫系统中被证明有很重要功能的microRNA关键靶基因。同时探究基因的UTR区microRNA结合位点在疾病,尤其是癌症和自身免疫病中的突变以及致病性较高的单核苷酸变异(SNV)[49],构建相关小鼠模型。这些研究可以更好地从遗传和生理学上研究microRNA的功能和作用机制。
5
microRNA研究的展望
5.1 靶基因介导的microRNA降解
TDMD, Target-directed miRNA degradation
microRNA降解是近年来新发现的microRNA调控机制,简单说是通过触发mRNA(trigger)诱导microRNA形成Ago-miRNA-trigger复合物,从而对可以结合到trigger mRNA上相应miRNA结合位点的microRNA进行降解。该机制最早由Zamore和Steitz实验室在2010年报道[50-51]。2020年Bartel和Mendell实验室进一步阐明降解机制依赖的泛素酶ZSWIM8和泛素化降解途径[52-53]。目前该机制已经被证明在包括果蝇和小鼠等多种动物体内存在,但其生理学意义和作用机制仍然有待进一步探究[54]。该机制可以通过含有特定miRNA结合位点的RNA来诱导体内microRNA降解,对理解基因调控的鲁棒性(robustness)很有价值。其调节microRNA在特定细胞组织中表达的功能也很有转化应用前景。
5.2 基于microRNA的疾病治疗
一些功能重要的microRNA,如miR-17~92、miR-155以及miR-122, 已经被证明参与了很多重要的器官发育和功能调控,甚至肿瘤和自身免疫病的发生发展。因此,很多公司和实验室在以microRNA为靶点,研究抑制microRNA表达和调控的手段。miR-122在肝脏组织中特异高表达,可以维持肝脏代谢功能稳态,在非酒精性脂肪肝炎和肝癌等疾病中扮演重要角色[55],也是首个被用于临床治疗的microRNA[56]。目前主要用反义核苷酸(ASO)来抑制microRNA功能,但是相关药物开发进展缓慢,在临床实验上结果不稳定。也有一些小分子化合物被发现可以调节microRNA表达水平,比如ABX464可以上调miR-124的表达而用于治疗类风湿性关节炎[57]。
此外,Disney实验室开发了一种利用小分子结合RNA并募集核酸酶切割或者降解RNA的方法(RIBOTAC),用于抑制microRNA前体的表达[58]。但是该方法还处于实验室研究和药物开发阶段,缺乏临床实验结果。无论是何种基于microRNA的治疗手段,都需要更多、更严谨、更耐心的研究。如前面所述,microRNA发挥作用的分子机制目前仍然没有完全厘清,尤其是针对microRNA调控细胞类型特异性的研究,仍需要构建相应动物模型,展开更多生理和分子水平上的实验[59]。
5.3 基于microRNA的合成生物学调控环路设计
microRNA剂量敏感的调控方式使其具备将靶基因表达水平控制在一个稳定范围的能力,非常符合合成生物学上需要保证信号输出(蛋白表达水平)精准稳定而且抗干扰能力强的设计思路。因此近年来一些合成生物学家开始将microRNA用于他们的合成环路设计中[60-61]。他们在要控制的基因的内含子插入microRNA并在该基因的3‘非编码区插入该microRNA的结合位点,形成一个反馈环路。并且经过不断尝试和改造,可以用于独立调控多个基因的表达水平。
从microRNA发现至今,已经有大量研究工作发表。但是此领域的研究依然方兴未艾,尤其是microRNA的分子机制研究,对其应用非常重要而且依然任重道远。microRNA研究也是一扇很好的窗口,引导科学家研究mRNA层面上的调控机制,对理解基因表达的精准调控并促进人类健康和发展有着深远的意义。
后记
本文根据作者的回忆和对本领域的理解写成。因为时间仓促,作者未对相关领域进行深入调研,文中难免会有主观、片面、和疏漏之处。并且许多实验室的重要工作也未在文中一一引用,在此表示歉意,并欢迎批评指正。
参考文献
[1]. Lee, R.C., Feinbaum, R.L., and Ambros, V.(1993). The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14. Cell 75, 843-854. 10.1016/0092-8674(93)90529-y.
[2]. Wightman, B., Ha, I., and Ruvkun, G.(1993). Posttranscriptional regulation of the heterochronic gene lin-14 by lin-4 mediates temporal pattern formation in C. elegans. Cell 75, 855-862. 10.1016/0092-8674(93)90530-4.
[3]. Pasquinelli, A.E., Reinhart, B.J., Slack, F., Martindale, M.Q., Kuroda, M.I., Maller, B., Hayward, D.C., Ball, E.E., Degnan, B., Muller, P., et al.(2000). Conservation of the sequence and temporal expression of let-7 heterochronic regulatory RNA. Nature 408, 86-89. 10.1038/35040556.
[4]. Reinhart, B.J., Slack, F.J., Basson, M., Pasquinelli, A.E., Bettinger, J.C., Rougvie, A.E., Horvitz, H.R., and Ruvkun, G.(2000). The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans. Nature 403, 901-906. 10.1038/35002607.
[5]. Ambros, V.(2008). The evolution of our thinking about microRNAs. Nat Med 14, 1036-1040. 10.1038/nm1008-1036.
[6]. Ambros, V.(2004). The functions of animal microRNAs. Nature 431, 350-355. 10.1038/nature02871.
[7]. Lagos-Quintana, M., Rauhut, R., Yalcin, A., Meyer, J., Lendeckel, W., and Tuschl, T.(2002). Identification of tissue-specific microRNAs from mouse. Curr Biol 12, 735-739. 10.1016/s0960-9822(02)00809-6.
[8]. Calin, G.A., Liu, C.G., Sevignani, C., Ferracin, M., Felli, N., Dumitru, C.D., Shimizu, M., Cimmino, A., Zupo, S., Dono, M., et al.(2004). MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci U S A 101, 11755-11760. 10.1073/pnas.0404432101.
[9]. Eis, P.S., Tam, W., Sun, L., Chadburn, A., Li, Z., Gomez, M.F., Lund, E., and Dahlberg, J.E.(2005). Accumulation of miR-155 and BIC RNA in human B cell lymphomas. Proc Natl Acad Sci U S A 102, 3627-3632. 10.1073/pnas.0500613102.
[10]. Lu, J., Getz, G., Miska, E.A., Alvarez-Saavedra, E., Lamb, J., Peck, D., Sweet-Cordero, A., Ebert, B.L., Mak, R.H., Ferrando, A.A., et al.(2005). MicroRNA expression profiles classify human cancers. Nature 435, 834-838. 10.1038/nature03702.
[11]. Chen, C.Z., Li, L., Lodish, H.F., and Bartel, D.P.(2004). MicroRNAs modulate hematopoietic lineage differentiation. Science 303, 83-86. 10.1126/science.1091903.
[12]. Tijsterman, M., Ketting, R.F., and Plasterk, R.H.(2002). The genetics of RNA silencing. Annu Rev Genet 36, 489-519. 10.1146/annurev.genet.36.043002.091619.
[13]. Bernstein, E., Kim, S.Y., Carmell, M.A., Murchison, E.P., Alcorn, H., Li, M.Z., Mills, A.A., Elledge, S.J., Anderson, K.V., and Hannon, G.J.(2003). Dicer is essential for mouse development. Nat Genet 35, 215-217. 10.1038/ng1253.
[14]. Koralov, S.B., Muljo, S.A., Galler, G.R., Krek, A., Chakraborty, T., Kanellopoulou, C., Jensen, K., Cobb, B.S., Merkenschlager, M., Rajewsky, N., and Rajewsky, K.(2008). Dicer ablation affects antibody diversity and cell survival in the B lymphocyte lineage. Cell 132, 860-874. 10.1016/j.cell.2008.02.020.
[15]. Muljo, S.A., Ansel, K.M., Kanellopoulou, C., Livingston, D.M., Rao, A., and Rajewsky, K.(2005). Aberrant T cell differentiation in the absence of Dicer. J Exp Med 202, 261-269. 10.1084/jem.20050678.
[16]. Cobb, B.S., Nesterova, T.B., Thompson, E., Hertweck, A., O'Connor, E., Godwin, J., Wilson, C.B., Brockdorff, N., Fisher, A.G., Smale, S.T., and Merkenschlager, M.(2005). T cell lineage choice and differentiation in the absence of the RNase III enzyme Dicer. J Exp Med 201, 1367-1373. 10.1084/jem.20050572.
[17]. Ota, A., Tagawa, H., Karnan, S., Tsuzuki, S., Karpas, A., Kira, S., Yoshida, Y., and Seto, M.(2004). Identification and characterization of a novel gene, C13orf25, as a target for 13q31-q32 amplification in malignant lymphoma. Cancer Res 64, 3087-3095. 10.1158/0008-5472.can-03-3773.
[18]. He, L., Thomson, J.M., Hemann, M.T., Hernando-Monge, E., Mu, D., Goodson, S., Powers, S., Cordon-Cardo, C., Lowe, S.W., Hannon, G.J., and Hammond, S.M.(2005). A microRNA polycistron as a potential human oncogene. Nature 435, 828-833. 10.1038/nature03552.
[19]. Xiao, C., Srinivasan, L., Calado, D.P., Patterson, H.C., Zhang, B., Wang, J., Henderson, J.M., Kutok, J.L., and Rajewsky, K.(2008). Lymphoproliferative disease and autoimmunity in mice with increased miR-17-92 expression in lymphocytes. Nat Immunol 9, 405-414. 10.1038/ni1575.
[20]. Ventura, A., Young, A.G., Winslow, M.M., Lintault, L., Meissner, A., Erkeland, S.J., Newman, J., Bronson, R.T., Crowley, D., Stone, J.R., et al.(2008). Targeted deletion reveals essential and overlapping functions of the miR-17 through 92 family of miRNA clusters. Cell 132, 875-886. 10.1016/j.cell.2008.02.019.
[21]. Rodriguez, A., Vigorito, E., Clare, S., Warren, M.V., Couttet, P., Soond, D.R., van Dongen, S., Grocock, R.J., Das, P.P., Miska, E.A., et al.(2007). Requirement of bic/microRNA-155 for normal immune function. Science 316, 608-611. 10.1126/science.1139253.
[22]. Thai, T.H., Calado, D.P., Casola, S., Ansel, K.M., Xiao, C., Xue, Y., Murphy, A., Frendewey, D., Valenzuela, D., Kutok, J.L., et al.(2007). Regulation of the germinal center response by microRNA-155. Science 316, 604-608. 10.1126/science.1141229.
[23]. Vigorito, E., Perks, K.L., Abreu-Goodger, C., Bunting, S., Xiang, Z., Kohlhaas, S., Das, P.P., Miska, E.A., Rodriguez, A., Bradley, A., et al.(2007). microRNA-155 regulates the generation of immunoglobulin class-switched plasma cells. Immunity 27, 847-859. 10.1016/j.immuni.2007.10.009.
[24]. Dorsett, Y., McBride, K.M., Jankovic, M., Gazumyan, A., Thai, T.H., Robbiani, D.F., Di Virgilio, M., Reina San-Martin, B., Heidkamp, G., Schwickert, T.A., et al.(2008). MicroRNA-155 suppresses activation-induced cytidine deaminase-mediated Myc-Igh translocation. Immunity 28, 630-638. 10.1016/j.immuni.2008.04.002.
[25]. Monticelli, S., Ansel, K.M., Xiao, C., Socci, N.D., Krichevsky, A.M., Thai, T.H., Rajewsky, N., Marks, D.S., Sander, C., Rajewsky, K., et al.(2005). MicroRNA profiling of the murine hematopoietic system. Genome Biol 6, R71. 10.1186/gb-2005-6-8-r71.
[26]. Xiao, C., Calado, D.P., Galler, G., Thai, T.H., Patterson, H.C., Wang, J., Rajewsky, N., Bender, T.P., and Rajewsky, K.(2007). MiR-150 controls B cell differentiation by targeting the transcription factor c-Myb. Cell 131, 146-159. 10.1016/j.cell.2007.07.021.
[27]. Kramer, N.J., Wang, W.L., Reyes, E.Y., Kumar, B., Chen, C.C., Ramakrishna, C., Cantin, E.M., Vonderfecht, S.L., Taganov, K.D., Chau, N., and Boldin, M.P.(2015). Altered lymphopoiesis and immunodeficiency in miR-142 null mice. Blood 125, 3720-3730. 10.1182/blood-2014-10-603951.
[28]. Boldin, M.P., Taganov, K.D., Rao, D.S., Yang, L., Zhao, J.L., Kalwani, M., Garcia-Flores, Y., Luong, M., Devrekanli, A., Xu, J., et al.(2011). miR-146a is a significant brake on autoimmunity, myeloproliferation, and cancer in mice. J Exp Med 208, 1189-1201. 10.1084/jem.20101823.
[29]. Kuchen, S., Resch, W., Yamane, A., Kuo, N., Li, Z., Chakraborty, T., Wei, L., Laurence, A., Yasuda, T., Peng, S., et al.(2010). Regulation of microRNA expression and abundance during lymphopoiesis. Immunity 32, 828-839. 10.1016/j.immuni.2010.05.009.
[30]. Li, Q.J., Chau, J., Ebert, P.J., Sylvester, G., Min, H., Liu, G., Braich, R., Manoharan, M., Soutschek, J., Skare, P., et al.(2007). miR-181a is an intrinsic modulator of T cell sensitivity and selection. Cell 129, 147-161. 10.1016/j.cell.2007.03.008.
[31]. Mok, Y., Schwierzeck, V., Thomas, D.C., Vigorito, E., Rayner, T.F., Jarvis, L.B., Prosser, H.M., Bradley, A., Withers, D.R., Martensson, I.L., et al.(2013). MiR-210 is induced by Oct-2, regulates B cells, and inhibits autoantibody production. J Immunol 191, 3037-3048. 10.4049/jimmunol.1301289.
[32]. Li, Y., Deng, X., Zeng, X., and Peng, X.(2016). The Role of Mir-148a in Cancer. J Cancer 7, 1233-1241. 10.7150/jca.14616.
[33]. Gonzalez-Martin, A., Adams, B.D., Lai, M., Shepherd, J., Salvador-Bernaldez, M., Salvador, J.M., Lu, J., Nemazee, D., and Xiao, C.(2016). The microRNA miR-148a functions as a critical regulator of B cell tolerance and autoimmunity. Nat Immunol 17, 433-440. 10.1038/ni.3385.
[34]. Bartel, D.P.(2009). MicroRNAs:target recognition and regulatory functions. Cell 136, 215-233. 10.1016/j.cell.2009.01.002.
[35]. Xiao, C., and Rajewsky, K.(2009). MicroRNA control in the immune system:basic principles. Cell 136, 26-36. 10.1016/j.cell.2008.12.027.
[36]. Alivernini, S., Gremese, E., McSharry, C., Tolusso, B., Ferraccioli, G., McInnes, I.B., and Kurowska-Stolarska, M.(2017). MicroRNA-155-at the Critical Interface of Innate and Adaptive Immunity in Arthritis. Front Immunol 8, 1932. 10.3389/fimmu.2017.01932.
[37]. Hsin, J.P., Lu, Y., Loeb, G.B., Leslie, C.S., and Rudensky, A.Y.(2018). The effect of cellular context on miR-155-mediated gene regulation in four major immune cell types. Nat Immunol 19, 1137-1145. 10.1038/s41590-018-0208-x.
[38]. Kang, S.G., Liu, W.H., Lu, P., Jin, H.Y., Lim, H.W., Shepherd, J., Fremgen, D., Verdin, E., Oldstone, M.B., Qi, H., et al.(2013). MicroRNAs of the miR-17 approximately 92 family are critical regulators of T(FH)differentiation. Nat Immunol 14, 849-857. 10.1038/ni.2648.
[39]. Jin, H.Y., Oda, H., Lai, M., Skalsky, R.L., Bethel, K., Shepherd, J., Kang, S.G., Liu, W.H., Sabouri-Ghomi, M., Cullen, B.R., et al.(2013). MicroRNA-17~92 plays a causative role in lymphomagenesis by coordinating multiple oncogenic pathways. EMBO J 32, 2377-2391. 10.1038/emboj.2013.178.
[40]. Lai, M., Gonzalez-Martin, A., Cooper, A.B., Oda, H., Jin, H.Y., Shepherd, J., He, L., Zhu, J., Nemazee, D., and Xiao, C.(2016). Regulation of B-cell development and tolerance by different members of the miR-17 approximately 92 family microRNAs. Nat Commun 7, 12207. 10.1038/ncomms12207.
[41]. Xie, J., Du, Y., Liu, D., Wu, J., Yang, K., He, X., Zhao, J., Hong, P., Liao, K., Zhang, H., et al.(2023). The miR-17 approximately 92 miRNAs promote plasma cell differentiation by suppressing SOCS3-mediated NIK degradation. Cell Rep 42, 112968. 10.1016/j.celrep.2023.112968.
[42]. Liao, K., Chen, P., Zhang, M., Wang, J., Hatzihristidis, T., Lin, X., Yang, L., Yao, N., Liu, C., Hong, Y., et al.(2024). Critical roles of the miR-17 approximately 92 family in thymocyte development, leukemogenesis, and autoimmunity. Cell Rep 43, 114261. 10.1016/j.celrep.2024.114261.
[43]. Jin, H.Y., Oda, H., Chen, P., Yang, C., Zhou, X., Kang, S.G., Valentine, E., Kefauver, J.M., Liao, L., Zhang, Y., et al.(2017). Differential Sensitivity of Target Genes to Translational Repression by miR-17~92. PLoS Genet 13, e1006623. 10.1371/journal.pgen.1006623.
[44]. Labi, V., Peng, S., Klironomos, F., Munschauer, M., Kastelic, N., Chakraborty, T., Schoeler, K., Derudder, E., Martella, M., Mastrobuoni, G., et al.(2019). Context-specific regulation of cell survival by a miRNA-controlled BIM rheostat. Genes Dev 33, 1673-1687. 10.1101/gad.330134.119.
[45]. Mukherji, S., Ebert, M.S., Zheng, G.X., Tsang, J.S., Sharp, P.A., and van Oudenaarden, A.(2011). MicroRNAs can generate thresholds in target gene expression. Nat Genet 43, 854-859. 10.1038/ng.905.
[46]. Chen, P., Liao, K., and Xiao, C.(2018). MicroRNA says no to mass production. Nat Immunol 19, 1040-1042. 10.1038/s41590-018-0215-y.
[47]. Vasudevan, S., Tong, Y., and Steitz, J.A.(2007). Switching from repression to activation:microRNAs can up-regulate translation. Science 318, 1931-1934. 10.1126/science.1149460.
[48]. Kato, M.(2023). Identify MicroRNA Targets Using AGO2-CLASH(Cross-linking, Ligation, and Sequencing of Hybrids)and AGO2-CLIP(Cross-Linking and Immuno-Precipitation)in Cells with or Without the MicroRNA of Interest Depleted. Methods Mol Biol 2666, 137-147. 10.1007/978-1-0716-3191-1_10.
[49]. Griesemer, D., Xue, J.R., Reilly, S.K., Ulirsch, J.C., Kukreja, K., Davis, J.R., Kanai, M., Yang, D.K., Butts, J.C., Guney, M.H., et al.(2021). Genome-wide functional screen of 3'UTR variants uncovers causal variants for human disease and evolution. Cell 184, 5247-5260 e5219. 10.1016/j.cell.2021.08.025.
[50]. Ameres, S.L., Horwich, M.D., Hung, J.H., Xu, J., Ghildiyal, M., Weng, Z., and Zamore, P.D.(2010). Target RNA-directed trimming and tailing of small silencing RNAs. Science 328, 1534-1539. 10.1126/science.1187058.
[51]. Cazalla, D., Yario, T., and Steitz, J.A.(2010). Down-regulation of a host microRNA by a Herpesvirus saimiri noncoding RNA. Science 328, 1563-1566. 10.1126/science.1187197.
[52]. Shi, C.Y., Kingston, E.R., Kleaveland, B., Lin, D.H., Stubna, M.W., and Bartel, D.P.(2020). The ZSWIM8 ubiquitin ligase mediates target-directed microRNA degradation. Science 370. 10.1126/science.abc9359.
[53]. Han, J., LaVigne, C.A., Jones, B.T., Zhang, H., Gillett, F., and Mendell, J.T.(2020). A ubiquitin ligase mediates target-directed microRNA decay independently of tailing and trimming. Science 370. 10.1126/science.abc9546.
[54]. Buhagiar, A.F., and Kleaveland, B.(2024). To kill a microRNA:emerging concepts in target-directed microRNA degradation. Nucleic Acids Res 52, 1558-1574. 10.1093/nar/gkae003.
[55]. Thakral, S., and Ghoshal, K.(2015). miR-122 is a unique molecule with great potential in diagnosis, prognosis of liver disease, and therapy both as miRNA mimic and antimir. Curr Gene Ther 15, 142-150. 10.2174/1566523214666141224095610.
[56]. Janssen, H.L., Reesink, H.W., Lawitz, E.J., Zeuzem, S., Rodriguez-Torres, M., Patel, K., van der Meer, A.J., Patick, A.K., Chen, A., Zhou, Y., et al.(2013). Treatment of HCV infection by targeting microRNA. N Engl J Med 368, 1685-1694. 10.1056/NEJMoa1209026.
[57]. Daien, C., Krogulec, M., Gineste, P., Steens, J.M., Desroys du Roure, L., Biguenet, S., Scherrer, D., Santo, J., Ehrlich, H., and Durez, P.(2022). Safety and efficacy of the miR-124 upregulator ABX464(obefazimod, 50 and 100 mg per day)in patients with active rheumatoid arthritis and inadequate response to methotrexate and/or anti-TNFalpha therapy:a placebo-controlled phase II study. Ann Rheum Dis 81, 1076-1084. 10.1136/annrheumdis-2022-222228.
[58]. Tong, Y., Lee, Y., Liu, X., Childs-Disney, J.L., Suresh, B.M., Benhamou, R.I., Yang, C., Li, W., Costales, M.G., Haniff, H.S., et al.(2023). Programming inactive RNA-binding small molecules into bioactive degraders. Nature 618, 169-179. 10.1038/s41586-023-06091-8.
[59]. Kilikevicius, A., Meister, G., and Corey, D.R.(2022). Reexamining assumptions about miRNA-guided gene silencing. Nucleic Acids Res 50, 617-634. 10.1093/nar/gkab1256.
[60]. Strovas, T.J., Rosenberg, A.B., Kuypers, B.E., Muscat, R.A., and Seelig, G.(2014). MicroRNA-based single-gene circuits buffer protein synthesis rates against perturbations. ACS Synth Biol 3, 324-331. 10.1021/sb4001867.
[61]. Du, R., Flynn, M.J., Honsa, M., Jungmann, R., and Elowitz, M.B.(2024). miRNA circuit modules for precise, tunable control of gene expression. bioRxiv. 10.1101/2024.03.12.583048.

















