数年前,TBtools 有一个功能,大体是可视化 Pfam 网站批量预测的序列保守结构域。后来的故事,大家大体都清楚,Pfam 一直出各种情况,要么就是 update 了格式,要么就是准备“倒闭关门”。最后呢,现在就是直接转交给 Interpro 去维护。于是呢,对应的功能我就干脆下线掉,因为没啥存在必要。
不过最近用了一段时间不同数据库,发现 pfam 的结果往往干净一点,简单来说,就是数据库的domain不多,但是还行,这个对于快速分析来说,还是实用。我测试了 Interpro 上 Pfam Search 的结果,认为还行。于是,干脆重新上线一个可视化功能,用于快速可视化对应结果,同时呢,配套这个推文。
使用 InterPro 网站进行PfamSearch
打开网页
https://www.ebi.ac.uk/interpro/
可以看到

点击「Search」,选择「By Sequence」
随后,选择本地文件上传,一般一次不超过100个蛋白序列,如超过,可以用 TBtools Fasta Split 先切分文件,分批处理。
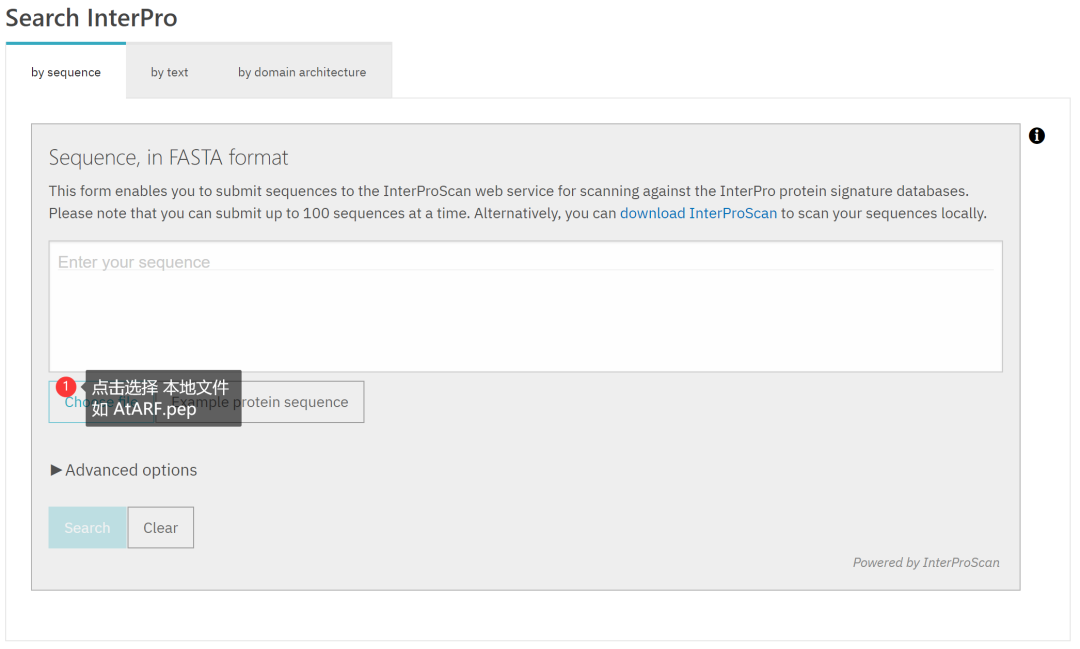
如果序列有终止密码子,会显示「Automatci FASTA clean up」,点击即可(你不点击也跑不了)
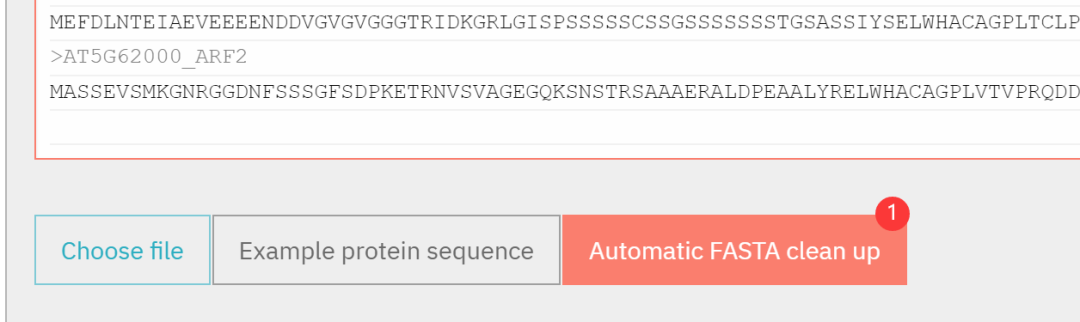
进入高级参数设置,设置仅扫描 Pfam
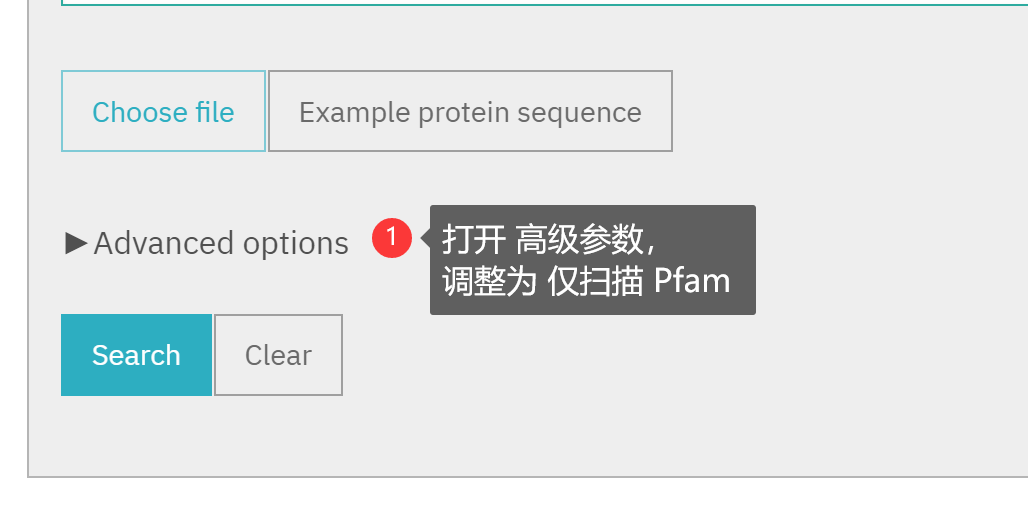
随后去掉所有默认勾选,手动勾选 Pfam,点击「Search」
会自动跳转到页面
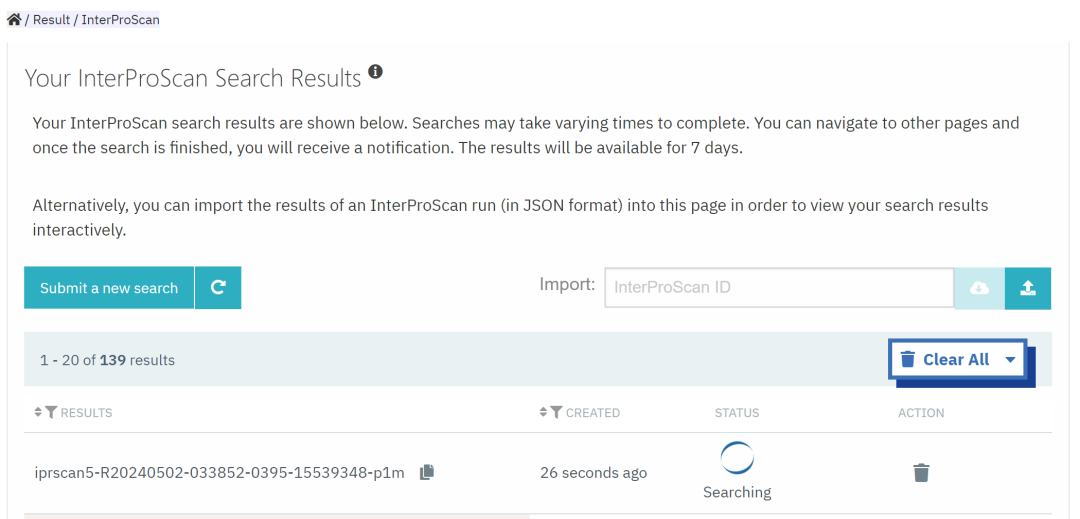
等待即可,一般是出去玩(数据库会自动记录IP,一段时间内可以找回结果的)。运行完成后,直接下载
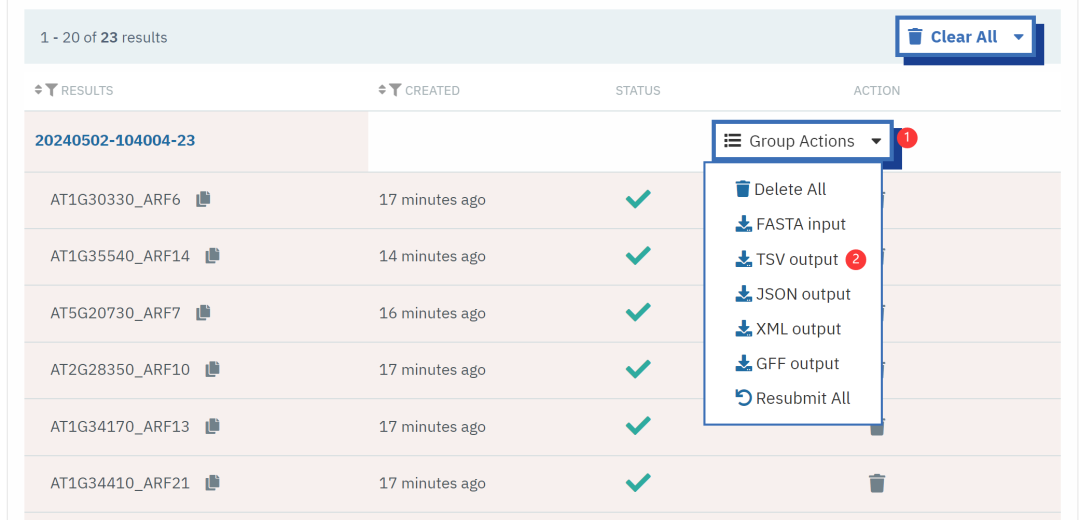
使用 TBtools 进行结果可视化
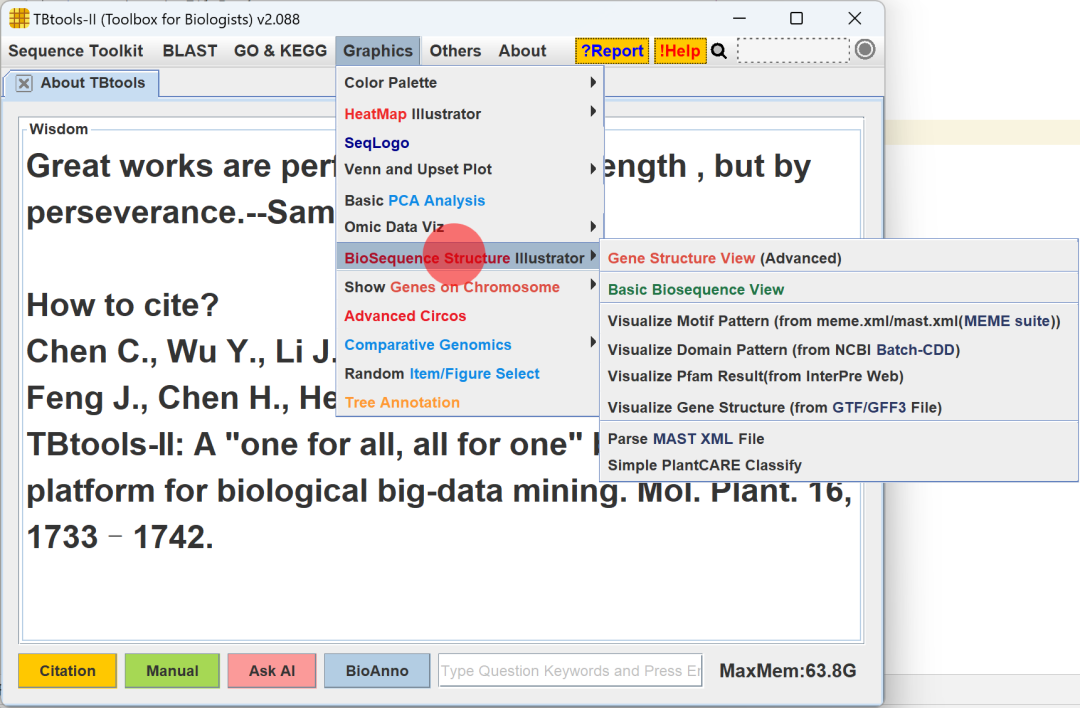
打开最新版本 TBtools,找到「Visualize Pfam Result...」
随后,直接放置待可视化的文件
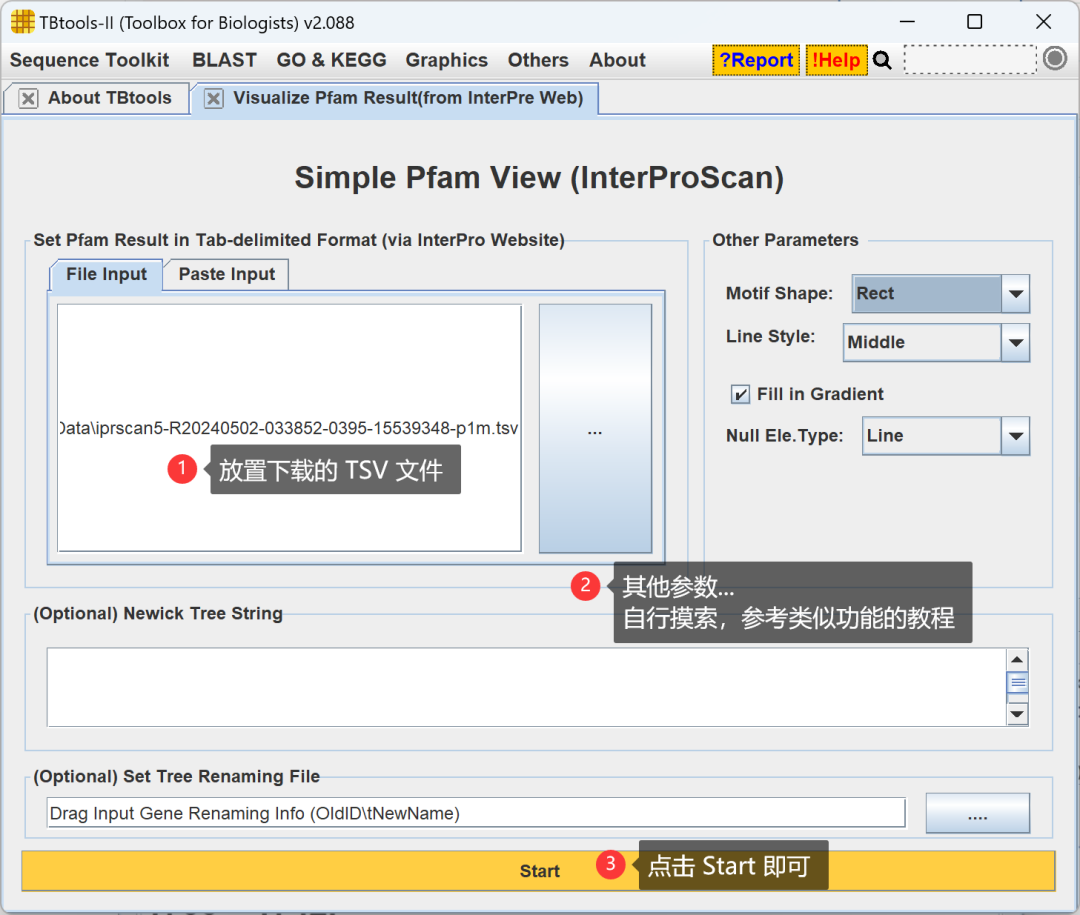
点击 Start 可以看到可视化结果
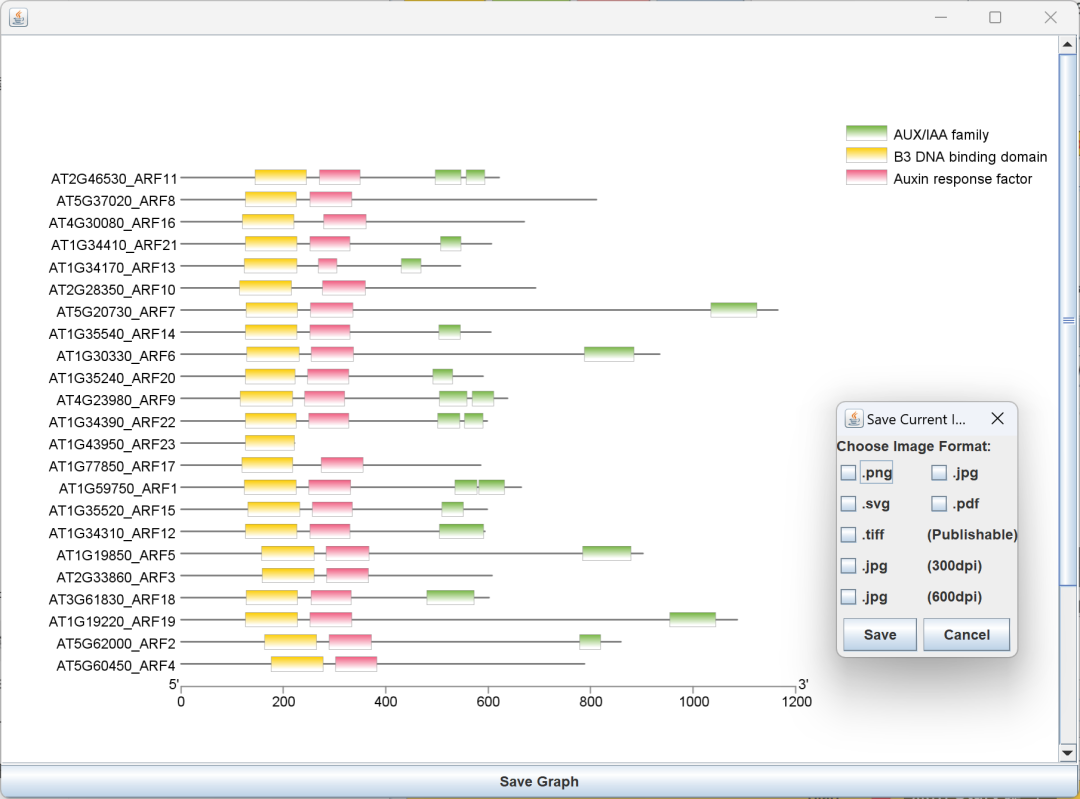
轻松搞定...
写在最后
Emmm,太久没写教程。写一下挺累~还是 coding 然后自己用来的舒服。不然每次要多花大半个小时。无论如何,祝大伙科研顺利~

















