一,简介
分子模拟是一种强大的计算工具,它可以通过模拟分子和原子的运动和相互作用来研究物质的结构和行为。这种方法在化学、物理学和生物学中具有广泛的应用,如研究蛋白质折叠、药物设计、材料科学和化学反应机制等。分子模拟的准确性在很大程度上依赖于所使用的模型和参数,而水模型在其中扮演了至关重要的角色。
水在自然界中无处不在,是生命活动和许多化学过程的重要介质。由于水的分子结构和氢键网络,使其具有独特的物理和化学性质,因此在分子模拟中准确地模拟水的行为显得尤为重要。然而,由于水分子间的复杂相互作用,以及水在不同环境中的不同表现,构建一个既能准确反映水的宏观性质又能精确描述微观行为的水模型一直是一个巨大的挑战。
本文旨在探讨水模型的起源与发展,以及溶剂化的基本原理。通过回顾水模型的发展历史,我们可以了解其演变过程和不同模型的特点与应用。此外,溶剂化作为一种重要的化学过程,广泛影响着溶质在溶剂中的行为,理解其原理对准确模拟生物分子和材料的行为至关重要。
二,水模型的起源与发展
2.1 早期水模型
Lennard-Jones模型[1]
Lennard-Jones模型是最早期的分子间相互作用模型之一,最初用于描述惰性气体原子的相互作用。该模型采用了Lennard-Jones势函数,定义了分子间的吸引和排斥力。尽管这种模型简单且计算效率高,但它无法准确捕捉水分子间复杂的氢键相互作用,因此在描述水的性质方面存在显著局限性。
Bernal-Fowler模型[2]
Bernal和Fowler在1933年提出了一种早期的水分子模型,尝试通过考虑水分子的四面体结构来解释其独特的物理性质。他们的模型首次引入了氢键的概念,虽然这一模型在技术上仍然有限,但为后续更复杂和精确的水模型奠定了基础。
2.2 经典水模型的出现
SPC(Simple Point Charge)模型[3]
SPC模型由Berendsen等人在1981年提出,是一种广泛使用的三点模型。该模型将水分子简化为三个带电点,其中两个点代表氢原子,一个点代表氧原子,且氢氧键长和HOH角固定。SPC模型能够在较低的计算成本下提供对液态水宏观性质的合理描述,但在一些细微性质上仍有不足。
TIP3P(Transferable Intermolecular Potential with 3 Points)模型[4]
TIP3P模型是由Jorgensen等人在1983年开发的三点水模型。与SPC模型相似,TIP3P模型同样将水分子简化为三个点电荷,但其参数经过优化,能够更好地再现水的密度和径向分布函数。TIP3P模型在分子动力学模拟中被广泛使用,尤其是在生物分子的研究中。
TIP4P[5]和TIP5P模型[6]
TIP4P模型由Jorgensen等人在1983年提出,是TIP3P模型的改进版。该模型增加了一个虚拟的M点,以更好地模拟水分子的电荷分布,从而更准确地描述水的静电性质。TIP5P模型进一步增加了两个虚拟点,来模拟氧原子上的孤对电子。这两个模型在模拟水的结构和动力学性质方面表现更好,但计算成本相对较高。
2.3 现代水模型的发展
POL3(Polarizable 3-site water model)[7]
POL3模型引入了极化效应,使得模型能够动态调整水分子的电荷分布。这种极化模型能够更准确地描述水在不同环境下的行为,特别是在强电场或界面情况下。
SPC/E(Extended Simple Point Charge)模型[8]
SPC/E模型是对SPC模型的扩展,由Berendsen等人在1987年提出。该模型在保持计算效率的同时,通过引入额外的能量修正项,提高了对水的密度,介电常数和自扩散系数的描述精度。
AMOEBA(Atomic Multipole Optimized Energetics for Biomolecular Applications)模型[9]
AMOEBA模型由Ponder等人开发,是一种多极子模型,能够准确地描述分子间复杂的电荷相互作用。该模型在模拟生物分子和溶液体系中表现出色,但计算复杂度较高。
通过这些水模型的发展,我们可以看到每个模型都是对前人工作的改进和优化,逐步提高了对水的复杂行为的模拟精度。尽管如此,开发一个能够在所有条件下都精确描述水的通用模型仍然是一个巨大的挑战。
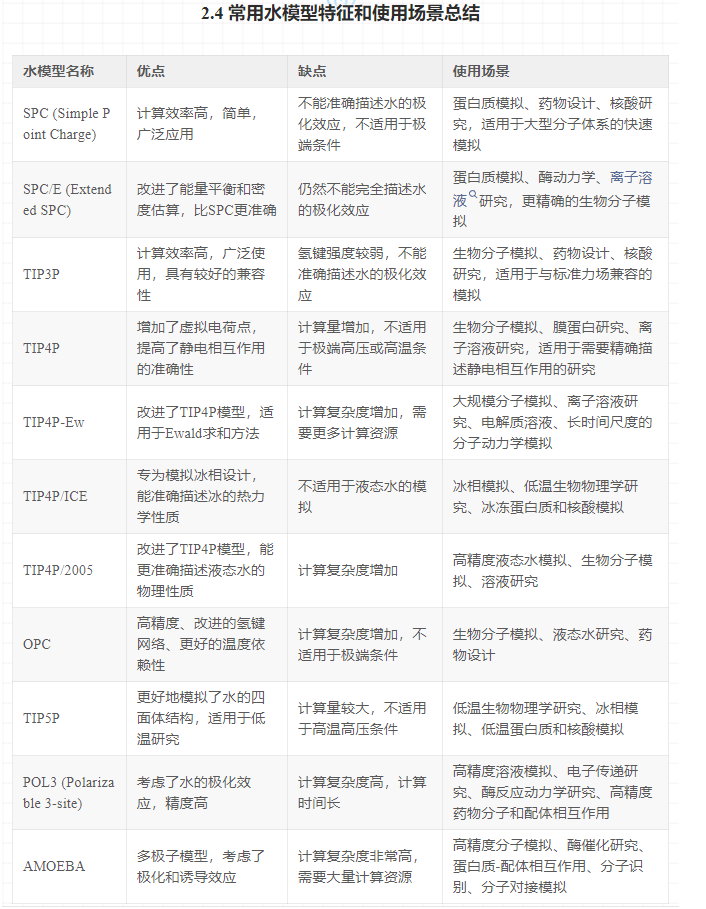
三,水模型的构建方法
3.1 参数化方法
水模型的构建通常涉及参数化过程,其中包括经验参数化和半经验参数化方法。
经验参数化
经验参数化方法依赖于实验数据和已知的分子性质。研究人员通过调整模型参数,使模拟结果与实验数据相吻合。例如,SPC和TIP3P模型的参数是通过拟合液态水的密度、静电性质和相变行为等实验数据得到的。
半经验参数化
半经验参数化方法结合了理论计算和实验数据。此方法利用简化的量子化学计算来获得初始参数,然后通过与实验数据对比进行调整。TIP4P和TIP5P模型就是使用这种方法优化得来的,以更好地模拟水分子的电荷分布和氢键网络。
3.2 基于量子化学计算的方法
量子化学计算方法在水模型的构建中也扮演了重要角色,特别是在极化模型和多极子模型的开发中。
从头计算
从头计算(Ab initio计算)是基于量子力学原理的第一性原理计算方法。通过从头计算,可以获得水分子的电子分布和势能面。POL3和AMOEBA模型利用从头计算得到的分子间相互作用信息来构建模型参数。
分子力场的构建
分子力场的构建涉及定义分子间相互作用的势函数,包括电荷分布、键长、键角和扭转角等。量子化学计算提供了这些参数的初始估计值,而后续的参数优化过程使模型能够更准确地反映实验观测到的物理性质。
3.3 实验数据的校准
实验数据在水模型的构建和验证过程中至关重要。
使用实验数据进行参数优化
水模型的参数通常需要通过与实验数据的对比进行校准。例如,水的介电常数、自扩散系数和热容等宏观性质是常用的校准标准。SPC/E模型就是通过引入能量修正项,使模型更好地再现水的介电常数。
模拟与实验结果的对比
构建好的水模型需要通过与实验结果的对比来验证其准确性。模拟结果应能够重现实验中观察到的水的物理和化学性质,如液态水的结构、动力学性质和相变行为。这种对比不仅验证了模型的准确性,还能指导进一步的模型改进。
通过上述方法,研究人员能够构建出越来越精确的水模型,以满足不同科学研究的需求。
四,溶剂化
4.1 溶剂化的定义
在详细探讨了水模型的起源与构建方法之后,我们接下来将重点介绍如何在分子模拟中应用这些水模型来进行溶剂化过程。溶剂化过程不仅是分子模拟中不可或缺的一部分,也是理解分子行为和相互作用的关键。通过溶剂化,研究人员能够在模拟中更真实地再现实际环境中的分子行为,从而提高模拟结果的准确性和可靠性。
溶剂化(Solvation)是指溶质分子在溶剂中被溶剂分子包围和稳定的过程。这个过程在化学、物理学和生物学中具有重要意义,因为许多化学反应、生物过程和材料行为都发生在溶液中。特别是在分子模拟中,通过对体系进行溶剂化,研究人员可以更准确地模拟生物分子、药物分子以及其他化学系统在实际环境中的行为。
4.2 溶剂化的重要性
溶剂化过程涉及溶剂分子围绕溶质分子形成溶剂壳层的过程。这个壳层通过各种分子间相互作用(如氢键、范德华力、电荷相互作用等)稳定溶质分子。例如,在水作为溶剂的情况下,水分子通过氢键和偶极相互作用,与溶质分子形成稳定的结构。这种溶剂化壳层不仅影响溶质分子的稳定性,还影响其动力学性质和反应机制。因此溶剂化过程对溶质分子的行为有着广泛的影响,在以下几个方面尤为重要:
稳定性与溶解性:溶剂化过程显著影响溶质分子的稳定性和溶解性。通过溶剂化,溶质分子能够在溶剂中保持稳定,防止聚集或沉淀。例如,蛋白质在水中的溶解和稳定性是由其与水分子的溶剂化相互作用决定的。
反应动力学:溶剂化还影响化学反应的动力学性质。溶剂分子通过稳定反应中间体和过渡态,降低反应活化能,从而加速反应速率。例如,酶促反应中,水分子不仅作为溶剂,还参与了酶与底物的相互作用和反应过程。
生物分子功能:生物大分子(如蛋白质、核酸等)在生理条件下的结构和功能依赖于溶剂化环境。水分子通过与生物大分子的氢键和疏水相互作用,影响其折叠、构象变化和功能表现。例如,蛋白质的活性位点常常包含一层特定排列的水分子,这些水分子在酶促反应中起着关键作用。
材料科学:在材料科学中,溶剂化过程影响材料的合成、加工和性能。例如,聚合物溶液的粘度和流变性质依赖于聚合物链与溶剂分子的相互作用。通过理解和控制溶剂化过程,可以优化材料的性能和应用。
药物设计与开发:在药物设计中,溶剂化对药物分子的吸收、分布、代谢和排泄(ADME)有重要影响。药物分子与水分子的相互作用影响其溶解度和生物利用度。通过分子模拟研究溶剂化过程,可以优化药物分子的结构,提高其药效和安全性。
综上所述,溶剂化过程在多种科学领域中具有重要的应用价值。通过分子模拟中的溶剂化操作,研究人员可以更好地理解和预测溶质分子在溶液中的行为,为科学研究和应用提供强有力的支持。
4.3 溶剂化的常见方法
在分子模拟中,有几种常见的方法用于对体系进行溶剂化:
盒子方法(Box Method)
盒子方法是最常用的溶剂化方法之一。在这种方法中,首先定义一个三维模拟盒,然后将溶质分子放置在盒子的中央,接着通过填充溶剂分子来完成溶剂化。
步骤:
定义模拟盒的尺寸:确定模拟盒的大小通常需要确保溶质分子与边界之间有足够的距离,以避免边界效应的影响。一般情况下,至少需要在溶质分子周围保留1.0纳米的空间。
放置溶质分子在盒子中心:将溶质分子(如蛋白质或小分子)放置在模拟盒的中心,以确保其被溶剂分子均匀包围。
添加溶剂分子:通过填充溶剂分子(如水分子)来填满整个模拟盒。通常使用预先平衡的溶剂模型(如SPC或TIP3P水模型)来进行填充。
优缺点:
优点:简单直接,易于实现,适用于大多数分子模拟系统。
缺点:可能会出现溶剂分子重叠或不均匀分布的问题,需要后续处理,如能量最小化和分子动力学平衡化。
随机放置法(Random Placement Method)
随机放置法是将溶剂分子随机地放置在模拟盒中。这种方法可以避免某些规则放置方法可能引入的偏差。
步骤:
定义模拟盒的尺寸:确定模拟盒的大小,以确保溶质分子与边界之间有足够的距离。
随机放置溶质分子:将溶质分子随机放置在模拟盒中,确保其中心位置合理。
随机放置溶剂分子:随机地将溶剂分子放置在模拟盒中的空隙中,避免与溶质分子发生重叠。
优缺点:
优点:避免了规则放置可能带来的系统误差,增加了溶剂分子的多样性和均匀性。
缺点:计算复杂度较高,可能需要更多的计算资源和时间来处理溶剂分子的随机分布。
预定义网格法(Predefined Grid Method)
预定义网格法是将模拟盒划分为规则的网格,然后将溶质和溶剂分子按照网格点进行放置。
步骤:
定义模拟盒的尺寸:确定模拟盒的大小,并将其划分为规则的网格。
划分网格:将模拟盒划分为一系列小的网格点,每个网格点代表一个潜在的溶剂分子放置位置。
按照网格点放置溶质和溶剂分子:将溶质分子放置在网格的中心位置,然后将溶剂分子按照剩余的网格点进行放置。
优缺点:
优点:均匀分布溶剂分子,减少重叠可能性,有助于快速生成初始配置。
缺点:需要仔细选择网格尺寸,以避免误差,且在高密度情况下可能不够灵活。
4.4 groamcs溶剂化方法及原理
这里以盒子方法为例子,简述一下gromacs常见的溶剂化体系的构建
示例1:构建纯水(溶剂)化体系的方法
步骤:
gmx solvate -cs spc216 -o water_box.gro -box 3 3 3
屏幕打印信息如下:
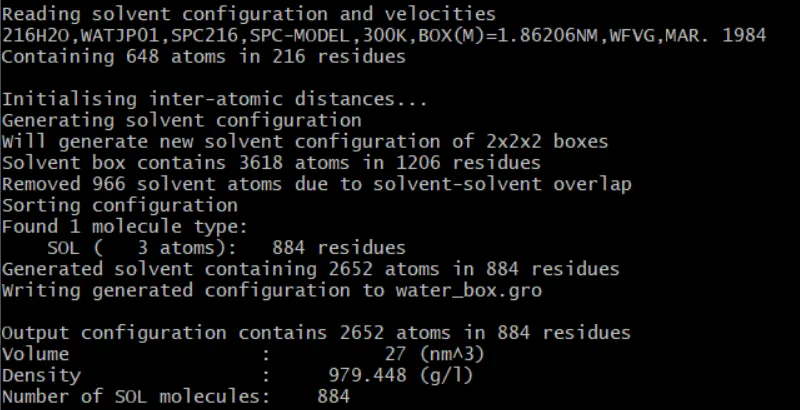
image-20240729161835494
Reading solvent configuration and velocities //程序正在读取溶剂的配置和速度信息。
216H2O,WATJP01,SPC216,SPC-MODEL,300K,BOX(M)=1.86206NM,WFVG,MAR. 1984://这行显示了预定义水盒子的详细信息。它表明使用的是216个水分子的SPC216模型,这是水分子的一个常见模型,适用于在300K(开尔文温度)下使用。BOX(M)表示原始水盒子的边长为1.86206纳米。
Containing 648 atoms in 216 residues //原始水盒子包含648个原子,分布在216个残基中。
Initialising inter-atomic distances... //程序正在初始化原子间的距离。
Generating solvent configuration //程序正在生成溶剂的配置。
Will generate new solvent configuration of 2x2x2 boxes //程序计划生成一个新的溶剂配置,其盒子尺寸是原始尺寸的2倍。(3nm/1.86206nm向上取整,XYZ三个方向,各填充2个sp216的预定于水盒子)
Solvent box contains 3618 atoms in 1206 residues //新的溶剂盒子包含3618个原子,分布在1206个残基中。
Removed 966 solvent atoms due to solvent-solvent overlap //由于溶剂分子之间的重叠,程序移除了966个溶剂原子。
Sorting configuration //程序正在对配置进行排序。
Found 1 molecule type:SOL(3 atoms):884 residues //程序发现了一种分子类型,即SOL(3个原子),共有884个残基。
Generated solvent containing 2652 atoms in 884 residues //生成的溶剂包含2652个原子,分布在884个残基中。
Writing generated configuration to water_box.gro //程序正在将生成的配置写入到water_box.gro文件中。
Output configuration contains 2652 atoms in 884 residues //输出的配置包含2652个原子,分布在884个残基中。
Volume :27(nm^3) //输出配置的体积为27立方纳米。
Density :979.448(g/l) //输出配置的密度为979.448克/升。
Number of SOL molecules:884 //输出配置中SOL分子的数量为884。
溶剂化的过程:如下图所示: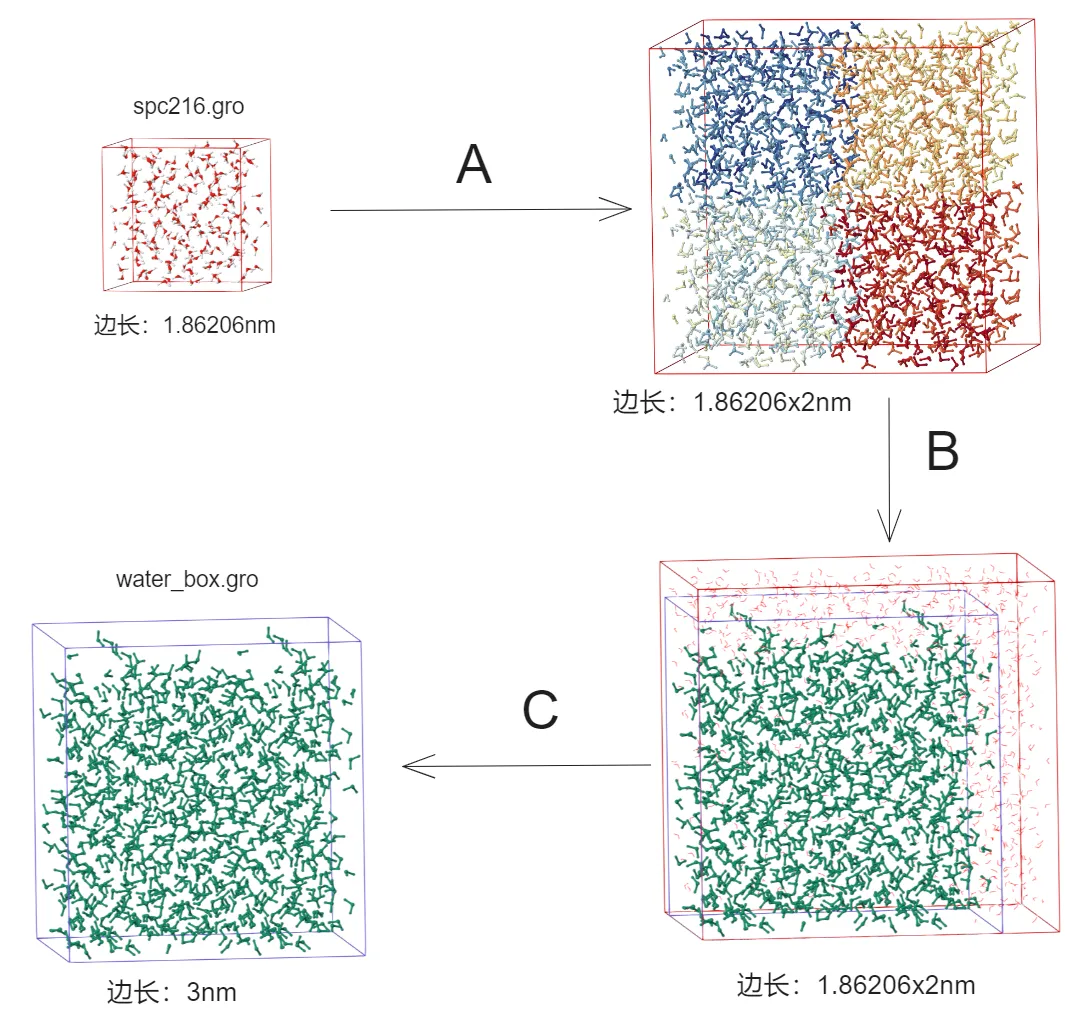
❝
GROMACS 提供几种预定义的水分子配置文件,用于快速设置溶液环境。最常用的预定义水盒子包括:
spc216.gro:这是一个小型的水分子盒子,通常用于SPC(Simple Point Charge)水模型。这种盒子包含216个水分子,排列成一个立方体结构。
tip4p.gro:用于TIP4P水模型的配置文件。包含216个水分子,排列成一个立方体结构。
tip5p.gro:用于TIP4P水模型的配置文件.包含512个水分子,排列成一个立方体结构。
这些预定义盒子通常是立方体或近似立方体的形状,因为立方体盒子最容易进行几何复制和处理。
示例2:构建溶质的(分子)溶剂化体系的方法
步骤如下:
(1).准备蛋白质分子,这里是使用的1ycr.pdb(PDBID:1YCR)的蛋白分子
(2)定义模拟盒:
使用editconf命令定义模拟盒的尺寸和形状。
gmx editconf -f 1ycr.pdb -o protein_box.gro -d 1.0 -bt cubic
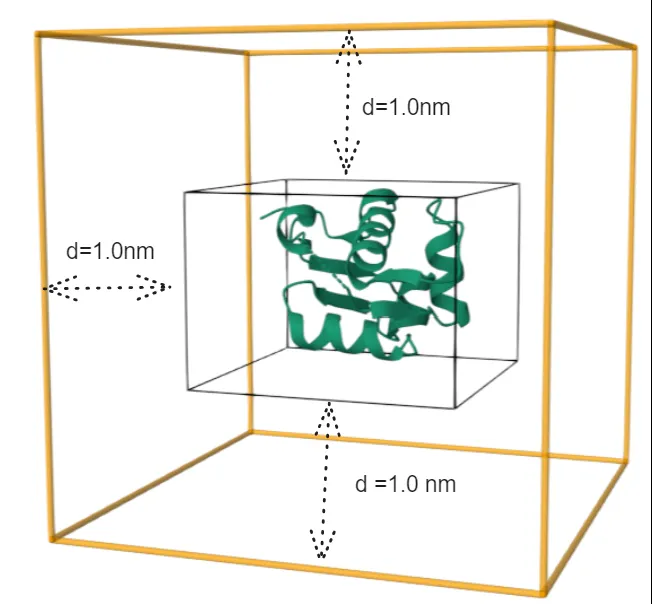
image-20240730143743113
❝
上面的命令是将蛋白质分子放置在盒子的中间,盒子的类型被定义为一个立方体(-bt cubic),并保持其距离盒子边缘1.0nm(-d 1.0)。到盒子边缘的距离(-d) 是一个重要的参数,因为在分子动力学模拟时,我们由于使用了周期性边界条件,因此需要满足最小图像约定,即溶质永远不应该"看到"其周期性图像。这里我们设置溶质(蛋白质分子)和盒子之间添加至少 1.0 nm 的距离 ,这意味着溶质分子的任何两个周期性图像之间至少有 2.0 nm(请注意,系统中的分子,例如蛋白质,可能会经历各种构象转变出现跨越周期性边界的情况)。对于模拟中常用的任何截止方案(范德华:0.8~1.2nm; 静电:1.0~1.6nm),这个距离应该足够了,但如果你使用的是自定义力场,则可能需要使用力场论文进行验证。
(3)添加溶剂分子:
使用solvate命令将水分子添加到模拟盒中。
gmx solvate -cs spc216 -cp protein_box.gro -o protein_solvate.gro
image-20240730153616818
溶剂化的过程:如下图所示:
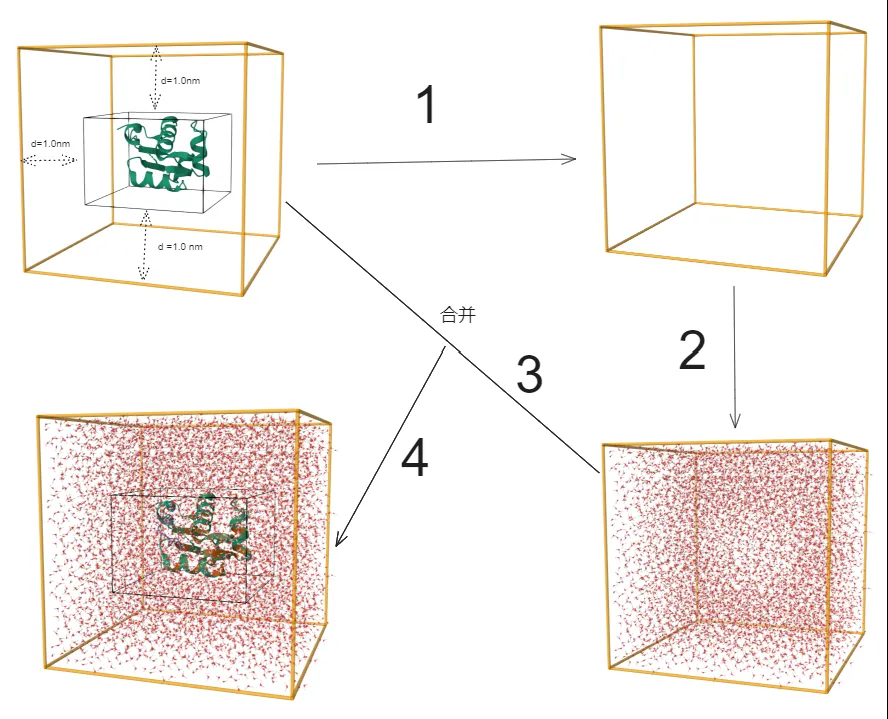
image-20240802153054166
gromacs构建溶剂化体系的原理
从上述的两个示例和溶剂化的过程中屏幕打印的信息,其实就可以知道大概知道gromacs的溶剂化的基本原理步骤的,更加详细的步骤可参见其关于solvate命令实现的源码:https://github.com/gromacs/gromacs/blob/2447fd95b93d00fd8472a52a6fada09eece2a42c/src/gromacs/gmxpreprocess/solvate.cpp#L861。这里简单概述的下gmx solvate核心功能逻辑的实现,如下:
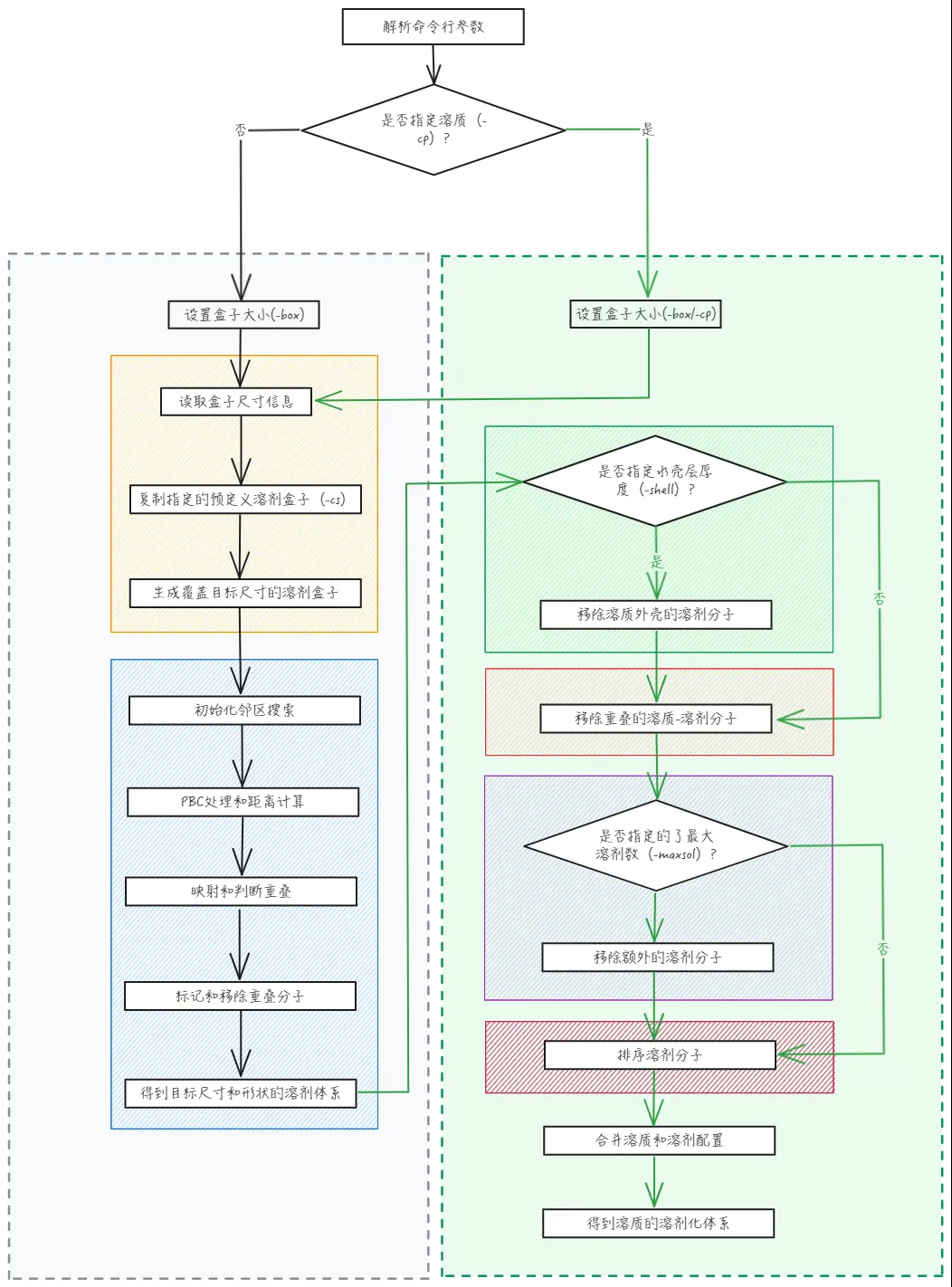
New Board(1)
gmx solvate 作为 GROMACS 软件中的一个功能强大的模块,它主要用于两个关键任务:生成溶剂盒子和进行溶质的溶剂化处理。
首先,它能够根据用户指定的参数,如溶剂文件(-cs)和盒子尺寸(-box),创建一个充满溶剂分子的盒子。这个功能特别适用于需要预设特定尺寸溶剂环境的情况。(上图左侧的灰色背景区域)
其次,该模块能够将一个溶质分子,比如蛋白质,溶解在溶剂中,形成溶剂化系统。用户需要指定溶质文件(-cp)和溶剂文件(-cs),程序会使用溶质坐标文件中预定义溶剂盒子,除非另外指定了盒子尺寸(-box)。(上图左侧灰色背景+右侧浅绿色背景区域)
在溶剂化过程中,gmx solvate 会智能地去除与溶质原子重叠的溶剂分子,确保生成的系统既符合物理实际,又适合后续的分子动力学模拟。
此外,它还提供了对溶剂分子进行排序的功能,以优化模拟的效率和准确性。整个模块的设计考虑了用户操作的简便性和模拟结果的可靠性,是 GROMACS 在分子模拟领域中不可或缺的工具之一。
下面将简述上述流程图中的关键执行两大类型溶剂化任务的核心功能函数:
1.图片:replicateSolventBox(),该函数用于复制溶剂盒子。它会根据目标和盒子的大小(-box)来计算预定义溶剂盒子(-cs spc216)在目标盒子各方向上的复制因子,然后再将预定溶剂盒子复制多次以填充目标盒子。这个过程中,可能会产生超出目标盒子的分子,因为目标盒子的尺寸可能无法完全被整数倍的盒子覆盖。比如示例1中情况
2.图片:removeSolventBoxOverlap(),该函数主要用于移除由于预定义的溶剂盒子和周期性边界条件(PBC)导致的重叠溶剂分子。简单来说其主要负责的任务是"剪裁",将replicateSolventBox()产生的溶剂化盒子"剪裁"为目标尺寸和形状的溶剂化体系。
❝
为什么需要"剪裁"?
1.目标盒子的尺寸可能无法完全被整数倍的预定义溶剂盒子覆盖。
2.预定义盒子是立方体,而目标盒子有可能是立方体,三斜晶系,截角八面体,菱形十二面体等。
为什么不直接移除超过目标尺寸边界的水分子?
1.预定义的水盒子模型在构建时已经考虑了周期性边界条件,因此它们被复制时堆叠成更大的溶剂化盒子时确保在边界处水分子的分布保持均匀,并且不会产生额外的密度变化,如果此时直接去掉边界外的水分子(如示例1中,蓝色框外的水分子)不考虑PBC下的重叠效应,可能会导致:
破坏系统的密度和均匀性:直接移除可能使得系统在边界处变得稀疏或不均匀,影响模拟结果的准确性。
忽略重要的物理现象:PBC下的分子可能出现在对侧边界,直接移除会忽略这种现象,导致模拟结果偏离真实物理情况。
removeSolventBoxOverlap()函数是如何做的?
具体过程见上述流程图,其主要做的事情就是对目标盒子的外的水分子进行PBC处理,将超出目标盒子范围的分子映射回盒子内,然后再通过距离来判断重叠的溶剂分子,并将其移除。
为什么需要像removeSolventBoxOverlap()函数这样?
保持系统的物理一致性:通过将所有分子映射到目标盒子内,确保在模拟中考虑到PBC效应,使得边界处的分子可以正确地相互作用。
避免不合理的分子分布:直接移除超出目标盒子边界的分子可能导致系统密度降低或边界效应的不准确表达。通过PBC处理和重叠检测,可以确保系统中的分子分布合理,模拟结果更接近实际物理情况
3.图片:removeSolventOutsideShell(),该函数根据给定的半径(-rshell)移除溶剂分子。它将去除所有距离溶质分子超过指定半径的溶剂分子。
❝
该过程同removeSolventBoxOverlap()中的过程类似,并不是直接移除水分子,而是考虑了PBC,这里不再赘述。
4.图片:removeSolventOverlappingWithSolute(),该函数用于移除与溶质分子重叠的溶剂分子。它通过检查溶质和溶剂分子之间的距离来识别和移除这些重叠的水分子。
❝
1.该过程同removeSolventBoxOverlap()中的过程类似,并不是直接移除水分子,而是考虑了PBC,这里不再赘述。
2.removeSolventBoxOverlap()和removeSolventOverlappingWithSolute()判断重叠的参数主要来源于-radius和-scale选项,以及数据文件vdwradii.dat中的范德华半径信息,程序优先读取vdwradii.dat中的范德华半径,并按照-scale缩放这些半径,对于对水中的蛋白质,使用默认值0.57可以得到接近1000 g/l的密度值。如果数据文件中找不到所需的半径值,相应的原子将通过-radius选项来设定距离。
5.图片:removeExtraSolventMolecules(),该函数用于移除额外的溶剂分子。如果实际溶剂分子数超过指定的最大值(max sol),它会随机移除多余的溶剂。
Reference
[1]
Lennard-Jones, J. E.(1924). On the determination of molecular fields. Proceedings of the Royal Society of London. Series A, Containing Papers of a Mathematical and Physical Character, 106(738), 463-477.:https://royalsocietypublishing.org/doi/10.1098/rspa.1924.0082
[2]
Bernal, J. D., & Fowler, R. H.(1933). A Theory of Water and Ionic Solution, with Particular Reference to Hydrogen and Hydroxyl Ions. The Journal of Chemical Physics, 1(8), 515-548.:https://pubs.aip.org/aip/jcp/article-abstract/1/8/515/177898/A-Theory-of-Water-and-Ionic-Solution-with?redirectedFrom=fulltext
[3]
Berendsen, H. J. C., Postma, J. P. M., van Gunsteren, W. F., & Hermans, J.(1981). Interaction models for water in relation to protein hydration. In Intermolecular Forces(pp. 331-342). Springer, Dordrecht.:https://link.springer.com/chapter/10.1007/978-94-015-7658-1_21
[4]
Jorgensen, W. L., Chandrasekhar, J., Madura, J. D., Impey, R. W., & Klein, M. L.(1983). Comparison of simple potential functions for simulating liquid water. The Journal of Chemical Physics, 79(2), 926-935.:https://pubs.aip.org/aip/jcp/article-abstract/79/2/926/776316/Comparison-of-simple-potential-functions-for?redirectedFrom=fulltext
[5]
orgensen, W. L., & Madura, J. D.(1983). Temperature and size dependence for Monte Carlo simulations of TIP4P water. Molecular Physics, 56(6), 1381-1392.:https://www.tandfonline.com/doi/abs/10.1080/00268978500103111
[6]
Mahoney, M. W., & Jorgensen, W. L.(2000). A five-site model for liquid water and the reproduction of the density anomaly by rigid, nonpolarizable potential functions. The Journal of Chemical Physics, 112(20), 8910-8922:https://pubs.aip.org/aip/jcp/article-abstract/112/20/8910/294328/A-five-site-model-for-liquid-water-and-the?redirectedFrom=fulltext
[7]
Caldwell, J., & Kollman, P.(1995). Structure and Properties of Neat Liquids Using Nonadditive Molecular Dynamics:Water, Methanol, and N‐Methylacetamide. The Journal of Physical Chemistry, 99(16), 6208-6219:https://pubs.acs.org/doi/10.1021/j100016a067
[8]
Berendsen, H. J. C., Grigera, J. R., & Straatsma, T. P.(1987). The missing term in effective pair potentials. The Journal of Physical Chemistry, 91(24), 6269-6271.:https://pubs.acs.org/doi/10.1021/j100308a038
[9]
Ponder, J. W., & Case, D. A.(2003). Force fields for protein simulations. Advances in Protein Chemistry, 66, 27-85.:https://pubmed.ncbi.nlm.nih.gov/14631816/

















